-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Technologie sekvenování nové generace: celogenomové, celoexomové a cílené – hotspot – sekvenování
Autoři: prof. RNDr. Slabý Ondřej, Ph.D.
Působiště autorů: Masarykův onkologický ústav, Brno ; CEITEC, Masarykova univerzita Brno
Vyšlo v časopise: Oborový speciál
Kategorie: Původní práce
Sekvenování nové generace (NGS, next generation sequencing) vykazuje oproti tradičním metodám používaným pro detekci genomových variant řadu výhod. Tradiční technologie, jako Sangerovo sekvenování, sekvenčně specifická PCR nebo fluorescenční in situ hybridizace (FISH), jsou vždy cílené pouze na konkrétní gen nebo variantu.
Základní principy sekvenování nové generace
Sekvenování nové generace (NGS, next generation sequencing) vykazuje oproti tradičním metodám používaným pro detekci genomových variant řadu výhod. Tradiční technologie, jako Sangerovo sekvenování, sekvenčně specifická PCR nebo fluorescenční in situ hybridizace (FISH), jsou vždy cílené pouze na konkrétní gen nebo variantu. V současné době, kdy je v onkologii často nezbytné paralelní vyšetřování většího množství genů/variant, kladou tyto konvenční metody vyšší nároky na množství biologického materiálu, často opakované biopsie a také delší čas do poskytnutí výsledků, protože jednotlivá vyšetření musejí být prováděna vždy nezávisle na sobě. Oproti tomu NGS umožňuje paralelní testování většího množství genů, případně celého exomu či genomu, a to i od většího množství pacientů v jednom experimentu. Tento přístup je spojen s nižšími nároky na množství vstupního biologického materiálu, a především s redukcí času do poskytnutí výsledků vyšetření, což má v onkologii zásadní význam.
NGS je založeno na masivním paralelním sekvenování mnoha fragmentů DNA. Tato technologie někdy bývá označována také jako vysokokapacitní sekvenování, hluboké sekvenování nebo sekvenování druhé generace. V současnosti již existuje řada technologií (nejčastěji používané Illumina a Ion Torrent) umožňujících diagnostickou aplikaci NGS. Napříč všemi platformami jsou zachovány základní principy NGS. Technologie NGS umožňuje sekvenovat DNA, a i když tři základní typy genomových variant –jednonukleotidové záměny (SNV), variabilita v počtu kopií genů (CNV) a krátké inzerce a delece (indels) – lze identifikovat na úrovni nádorové DNA, neméně důležité fúzní geny detekujeme pomocí analýzy nádorové RNA. Na jednu kopii fúzního genu v nádorovém genomu totiž připadají řádově stovky až tisíce kopií jeho RNA transkriptu. Detekcí fúzních genů na úrovni RNA proto ve srovnání s DNA dosahujeme mnohonásobně vyšší citlivosti. Před vlastní sekvenační reakcí je ovšem nezbytné provést přepis (reverzní transkripci) nádorové RNA do cDNA.
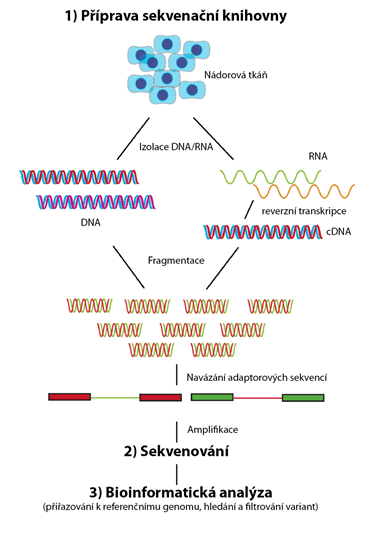
Prvním krokem NGS je příprava vzorku DNA pro účely sekvenace neboli příprava tzv. sekvenační knihovny. V tomto kroku je vždy zahrnuta izolace DNA ze vzorku nádoru (případně přepis nádorové RNA do cDNA), fragmentace DNA, navázání tzv. adaptorových sekvencí na konce fragmentů DNA, což umožní jejich globální namnožení (amplifikaci), a dále jejich fixaci na solidní povrch. Následně, ve druhém kroku, dochází k vlastní sekvenaci syntézou. K imobilizovaným fragmentům DNA jsou komplementárně připojovány jednotlivé nukleotidy (ACTG), což je spojeno s emisí vždy jiného fyzikálního signálu zachycovaného detektorem. Takto získané sekvence jednotlivých fragmentů jsou ve třetím kroku bioinformaticky analyzovány. V první fázi jsou získané sekvence přiřazovány (alignment) k referenčnímu genomu či sekvenci považované za sekvenci referenční. Následně probíhá analýza, jejímž cílem je nalézt varianty v sekvenovaném vzorku DNA (variant calling), poté anotace nalezených variant a dále nejsložitější krok bioinformatické analýzy, kterým je filtrování nalezených variant. Zde jsou postupně eliminovány varianty podle různých kritérií, v první fázi automaticky (například dle schopnosti varianty měnit strukturu kódovaného proteinu, tzv. nesynonymní varianty) a následně manuálně (například filtrování podle klinické evidence). Cílem je zúžit seznam nalezených variant na takové, které jsou biologicky relevantní v kontextu daného nádorového onemocnění (driverové mutace) a optimálně také klinicky relevantní (actionable mutace) s jasným dopadem pro terapeutické plánování. Workflow technologie sekvenování nové generace je shrnuto na obr. 1.
Obr. 1 Využití technologie sekvenování nové generace pro studium nádorové DNA.
Představíme-li si konkrétní pozici v lidském genomu, resp. genu, např. BRAF V600E (v sekvenci genu pro BRAF to odpovídá pozici a záměně c. 1799T > A), počet fragmentů DNA získaných z nádorového vzorku topologicky pokrývajících tuto pozici určuje, kolikrát bude daná pozice genu v sekvenační reakci tzv. přečtena, tedy jaká bude v dané pozici hloubka čtení (sequencing depth), resp. jaké zde bude pokrytí neboli coverage.
Představme si, že máme biologický vzorek tvořený tisícem buněk, ze kterých je 50 % nádorových. Z těchto 500 nádorových buněk 400 buněk vykazuje heterozygotní stav pro aktivační mutaci BRAF V600E – to je 40 % všech buněk obsahuje mutaci v jedné alele, tzn. 20 % DNA ve vzorku nese mutaci. Pokud danou pozici při sekvenaci přečteme s hloubkou čtení 20X, teoreticky 4 z 20 čtení zachytí mutovanou alelu A, 16 z 20 čtení potom nemutovanou alelu T.Základní aplikace sekvenování nové generace
Za dvě základní aplikace NGS lze považovat (i) sekvenaci genomové DNA nebo jejích částí s cílem identifikovat SNV, CNV a indels a dále (ii) sekvenaci transkriptomu, tzv. RNAseq; technicky se jedná o sekvenaci DNA získané přepisem nádorové RNA s cílem identifikovat přítomnost fúzních genů. Z hlediska genomové DNA lze potom sekvenovat zárodečnou DNA (germinální varianty), získanou obvykle z periferních leukocytů či bukálního stěru, obyčejně pro účely klinické genetiky, případně DNA nádorovou (somatické varianty), získanou izolací ze vzorku nádorové tkáně pro účely terapeutického plánování.
Z hlediska tzv. šíře pokrytí (breadth of coverage) neboli procenta genomu pokrytého sekvenační reakcí dělíme základní aplikace NGS na (i) celogenomové sekvenování (WGS, whole genome sequencing), (ii) celoexomové sekvenování (WES, whole exome sequencing) a (iii) cílené (hotspot) sekvenování, založené na použití tzv. sekvenačních panelů (srovnání v tabulce 1).
Tabulka 1 – Srovnání základních aplikací sekvenování nové generace.
Aplikace NGS
Popis
Výhody
Nevýhody
Celogenomové sekvenování (WGS)
Stanovení kompletní sekvence genomové DNA
- Poskytuje sekvence kódujících i nekódujících oblastí genomu
- Představuje nejkomplexnější způsob charakterizace genomové DNA
- Velký význam z hlediska translačního onkologického výzkumu
- Umožňuje výpočet mutační nálože (TMB)
- Velké množství získaných dat klade vysoké nároky na jejich interpretaci, a to především v případě náhodných potenciálně rizikových nálezů
- Finanční a časová náročnost
Celoexomové sekvenování (WES)
Stanovení sekvence přibližně 1 % genomové DNA představující kódující oblasti
- Většina
patogenních variant (85 %) leží v kódujících částech genomu
- Funkční dopady těchto variant jsou lépe interpretačně uchopitelné
- Oproti WGS umožňuje větší hloubku čtení
- Umožňuje výpočet mutační nálože (TMB)
- Neumožňuje identifikaci variant v nekódujících oblastech genomu
- Problém s náhodnými potenciálně rizikovými nálezy
Cílené (hotspot) sekvenování (sekvenační panely)
Stanovení sekvence vybraných genů, případně vybraných genomových oblastí
- Méně finančně a časově náročné
- Cílení na konkrétní geny a varianty (hotspoty) umožňuje jejich snadnou klinickou interpretaci
- Malé riziko náhodných nálezů
- Vysoká senzitivita
- Metodika umožňuje snadnější optimalizaci a standardizaci ve srovnání s WGS a WES
- Umožňuje výpočet mutační nálože TMB (při počtu genů na panelu vyšším než 200)
- Největší limitací je nízká šíře pokrytí, determinovaná počtem sekvenovaných genů, která může vést k nezachycení potenciálně klinicky významné varianty (s rostoucím počtem genů pokrytých panelem tato limitace úměrně ztrácí na významu)
Celogenomové sekvenování umožňuje detekovat všechny typy variant napříč celým genomem, v kódujících (tzv. exom) i nekódujících oblastech, a tak umožňuje identifikovat i nové, dříve nepopsané nádorové varianty. Srovnání zárodečného genomu s genomem nádorovým poskytne skutečně komplexní pohled na specifické změny přítomné v nádorovém genomu. Nicméně množství dat získané pomocí WGS klade vysoké nároky na jejich interpretaci, a to především v případě náhodných potenciálně rizikových, ale těžce interpretovatelných nálezů.
Celoexomové sekvenování svojí šíří pokrývá pouze kódující oblasti genomu, tedy přibližně 1 % z jeho celkové sekvence. Nicméně přibližně 85 % patogenních variant se nachází právě v kódujících oblastech, což činní výstupy WES oproti WGS jednodušší z hlediska interpretace. Nižší šíře pokrytí WES umožňuje oproti WGS větší hloubku čtení, a tak i vyšší senzitivitu pro záchyt variant v heterogenním vzorku nádorové tkáně. Z hlediska klinicky významného informačního obsahu představuje WES velice dobrou alternativu k WGS.
V současnosti je pro účely precizní onkologie nejvíce používáno cílené (hotspot) sekvenování, založené na tzv. sekvenačních panelech. V rámci tohoto přístupu je sekvenován předem definovaný panel řádově desítek až stovek genů významných v nádorové biologii, případně pro terapeutické plánování. Tento přístup má oproti WGS a WES řadu výhod především z praktického hlediska. Ještě menší šíře pokrytí, než má WES, umožňuje větší hloubku čtení, a tedy nejvyšší senzitivitu, což může mít svůj význam především u nádorových biopsií s nízkým zastoupením nádorových buněk. Sekvenační panely jsou méně finančně nákladné, jsou jednodušší z hlediska interpretace a nevedou k náhodným nálezům. Z hlediska technologického umožňují panely lepší optimalizaci sekvenačního protokolu a vyšší míru jeho standardizace. Mezi komerčně nabízenými sekvenačními panely se setkáme s panely obsahujícími jak desítky, tak i stovky genů, přičemž s narůstajícím počtem genů zařazených do panelu logicky narůstá také pravděpodobnost záchytu klinicky relevantní mutace a rovněž přesnost výpočtu tzv. mutační nálože nádoru – viz níže. Na druhou stranu, cílené (hotspot) sekvenování nikdy nepokrývá všechny potenciálně klinicky relevantní genomové varianty, a pravděpodobnost jejich záchytu pomocí WGS a WES bude proto vždy vyšší. Informace o individuálních nádorových exomech či genomech má rovněž významnou hodnotu pro translační výzkum v onkologii.
Ať již použijeme kteroukoliv z výše uvedených aplikací genomového sekvenování, je možné její výstupy použít pro výpočet tzv. mutační nálože nádoru neboli TMB (tumor mutational burden). V případě sekvenačních panelů lze výpočet TMB provést pouze v případě, že panel pokrývá 200 a více genů. Teprve od tohoto limitního počtu genů začíná hodnota TMB získaná ze sekvenačního panelu korelovat s TMB získanou pomocí WGS a WES. TMB je definována jako počet nesynonymních (protein měnících) mutací na 1 Mb (na milion bází) DNA a jeho hodnota může sloužit jako citlivý ukazatel odpovědi na protinádorovou imunoterapii.
Role sekvenování nové generace ve fungování molekulárněonkologické indikační komise
Použitím jakékoliv z výše uvedených aplikací NGS získáme více či méně komplexní informace o individuálním nádorovém genomu. Tyto informace jsou použity jako podklad pro přípravu detailních výsledkových protokolů obsahujících přehled klinicky relevantních variant a stupeň klinické evidence jejich vztahu ke konkrétnímu preparátu a léčebné odpovědi. Takto připravené protokoly jsou následně projednávány v kontextu celkového klinického stavu pacienta na multioborové molekulárněonkologické indikační komisi (MTB, molecular tumor board) s cílem vytvoření individuálního terapeutického plánu založeného na biologických vlastnostech daného tumoru.
Literatura
- Moorcraft S. Y., Gonzalez D., Walker B. A. Understanding next generation sequencing in oncology: A guide for oncologists. Crit Rev Oncol Hematol. 2015; 96 (3): 463–74.
- Strom S. P. Current practices and guidelines for clinical next-generation sequencing oncology testing. Cancer Biol Med. 2016 Mar; 13 (1): 3–11.
- Carter T. C., He M. M. Challenges of Identifying Clinically Actionable Genetic Variants for Precision Medicine. J Healthc Eng. 2016; doi: 10.1155/2016/3617572.
prof. RNDr. Ondřej Slabý, Ph.D.
Vedoucí výzkumné skupiny
CEITEC, Masarykova Univerzita
Brno
&
Vědecký tajemník
Masarykův onkologický ústav
Žlutý kopec 7
Brno
Štítky
Detská onkológia Detská rádiológia Chirurgia všeobecná Interné lekárstvo Kardiochirurgia Kardiológia Onkológia Rádiodiagnostika Rádioterapia
Článok vyšiel v časopiseOborový speciál
Najčítanejšie tento týždeň
- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Kombinace metamizol/paracetamol v léčbě pooperační bolesti u zákroků v rámci jednodenní chirurgie
- Antidepresivní efekt kombinovaného analgetika tramadolu s paracetamolem
- Parazitičtí červi v terapii Crohnovy choroby a dalších zánětlivých autoimunitních onemocnění
-
Všetky články tohto čísla
- CD8+ tumor infiltrující lymfocyty jako prognostický faktor high-grade serózních karcinomů ovaria
- Riziko vzniku karcinomu prsu, ovarií a kontralaterálního karcinomu prsu u nositelek mutací BRCA1 a BRCA2
- Užívání dlouhodobě působících inzulinových analog a incidence karcinomu prsu
- Optimální terapie časného stadia karcinomu prsu – výsledky osmiletého sledování
- Receptor HER2 jako maják – trastuzumab-emtansin je účinný u těžce předléčených pacientek s karcinomem prsu
- Pertuzumab v léčbě HER2-pozitivního karcinomu prsu zlepšuje přežití bez známek invazivního onemocnění
- Triptorelin zmírňuje příznaky dolních cest močových u pacientů s karcinomem prostaty
- Volba biologického léku v 1. linii léčby pokročilého/metastatického kolorektálního karcinomu s divokým typem KRAS
- Potenciál afatinibu v radiosenzitizaci HNSCC
- Oxid arzenitý v léčbě APL umožňuje redukci kumulativní dávky antracyklinů
- Onkologický screening u pacientů s nevyprovokovanou žilní trombózou
- Využívání metod alternativní medicíny u pacientů hematologické a onkologické kliniky
- Vemurafenib v léčbě generalizovaného melanomu - kazuistika
- Technologie sekvenování nové generace: celogenomové, celoexomové a cílené – hotspot – sekvenování
- Mediální onkologická realita
- Imunoterapie nádorových onemocnění
- Pro úspěšnost kombinované imunoterapie v léčbě nádorů je rozhodující pořadí aplikace jednotlivých protilátek
- Oborový speciál
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Technologie sekvenování nové generace: celogenomové, celoexomové a cílené – hotspot – sekvenování
- Pro úspěšnost kombinované imunoterapie v léčbě nádorů je rozhodující pořadí aplikace jednotlivých protilátek
- Riziko vzniku karcinomu prsu, ovarií a kontralaterálního karcinomu prsu u nositelek mutací BRCA1 a BRCA2
- Vemurafenib v léčbě generalizovaného melanomu - kazuistika
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



