-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
DNA a mikroRNA čipové technologie v diagnostice a predikci u pacientů s renálním karcinomem
DNA and MicroRNA Microarray Technologies in Diagnostics and Prediction for Patients with Renal Cell Carcinoma
Renal cell carcinoma accounts for approximately 3% of adult cancers and has the highest lethality of urological malignancies. Research focusing on carcinogenesis and development of renal cell carcinoma has led to the identification of the key signalling pathways and consequently targeted cancer therapy which improves time to progression or overall survival of renal cell carcinoma patients. Today, microarray technologies are some of the most efficient methods used in gene expression studies. Through one microarray experiment we can simultaneously determine the expression of thousands of genes, thus facilitating research of examined biological models. The most frequently used of the microarray technologies are DNA microarrays enabling global analysis of the mRNA (messenger RNA) expression, while recently, microarray platforms modified to detect short non coding RNAs (microRNAs) have been employed (microRNA microarrays). MicroRNAs significantly affect the behaviour of tumour cells by post transcriptional regulation of the gene expression. In the research into renal cell carcinoma, microarray technologies have been applied in more than twenty studies over the past five years. These papers describe the potential of microarrays to distinguish tumour tissue from normal renal parenchyma, to classify renal cell carcinomas according to histological subtypes, to identify expression profiles predicting metastasizing in primary renal tumours, and to determine the prognosis of particular renal cell carcinoma patients. The aim of this review is to summarize the results from microarray studies of renal cell carcinoma realized to date and to present their potential usage in diagnostic and therapeutic protocols.
Key words:
renal cell carcinoma – DNA microarrays – microRNA microarrays – prognosis – prediction
Autoři: O. Slabý 1; M. Svoboda 1
; J. Michálek 2; R. Vyzula 1
Působiště autorů: Klinika komplexní onkologické péče, Masarykův onkologický ústav, Brno2 Univerzitní centrum buněčné imunoterapie, Masarykova univerzita, Brno 1
Vyšlo v časopise: Klin Onkol 2009; 22(5): 202-209
Kategorie: Přehledy
Souhrn
Renální karcinom tvoří přibližně 3% zhoubných nádorů dospělé populace a z urologických malignit dosahuje nejvyšší letality. Výzkum v oblastech vzniku a vývoje renálního karcinomu vedl k identifikaci klíčových signálních drah a následně i cílené protinádorové léčby prodlužující čas do progrese onemocnění, případně i celkové přežití léčených pacientů. Mikročipové technologie patří v současnosti mezi nejefektivnější metody studia genové exprese. Pomocí jednoho čipu lze paralelně detekovat expresi až desítek tisíc genů, a výrazně tak urychlit výzkum studovaných biologických modelů. K nejčastěji používaným mikročipům patří DNA čipy analyzující expresi mediátorové RNA (mRNA), nově se začínají uplatňovat mikročipové platformy detekující krátké nekódující RNA (mikroRNA), tzv. mikroRNA čipy. MikroRNA post transkripčně regulují genovou expresi, a tak zásadním způsobem ovlivňují vlastnosti buňky. Ve výzkumu renálního karcinomu byly za posledních pět let využity mikročipové technologie ve více než dvaceti studiích. Tyto práce popisovaly schopnost mikročipů odlišit nádorovou tkáň od normálního renálního parenchymu, klasifikovat zhoubné novotvary ledviny podle histologických podtypů, identifikovat genové profily predikující metastazování renálního karcinomu a determinující prognózu jednotlivých pacientů. Cílem tohoto přehledu je shrnout výsledky z dosud provedených mikročipových studií u renálního karcinomu a prezentovat jejich potenciální uplatnitelnost v diagnostických a léčebných postupech.
Klíčová slova:
renální karcinom – DNA čipy – mikroRNA čipy – prognóza – predikceÚvod
Přestože zhoubné novotvary (ZN) ledvin představují u dospělých přibližně pouze 3% ze všech nově diagnostikovaných maligních nádorů, jejich incidence narůstá nepřetržitě od 70. let minulého století. Ve srovnání s výskytem ZN ledviny s ostatními zeměmi světa se Česká republika v roce 2002 zařadila na první místo. Roční incidence a mortalita ZN ledviny v České republice v posledních letech opakovaně překročila 2 800, resp. 1 200 případů, což představuje incidenci přibližně 28 a mortalitu 12 případů na 100 000 obyvatel [1]. V celkové incidenci ZN ledvin převažují v ČR muži nad ženami v poměru 1,5–1,7 : 1, u renálního karcinomu může být rozdíl ještě výraznější (až 2 : 1).
V ledvinách se mohou vyskytnout nádory různé histogeneze. Přibližně 85% tvoří karcinomy vycházející z epitelu v oblasti parenchymu ledviny, cca po 5% představují uroteliální karcinomy z epitelu ledvinné pánvičky a embryonální nádory, zbývající část připadá na sarkomy, lymfomy a některé další vzácné podjednotky. Z karcinomů jednoznačně převažuje renální karcinom ze světlých buněk (80% karcinomů), vycházející z buněk proximálních tubulů (clear cell carcinoma – cRCC). Z proximálních tubulů vzniká i papilární renální karcinom (pRCC) (10% karcinomů), z distálních tubulů vychází chromofobní renální karcinom (chRCC) (5% karcinomů). Vzácně se vyskytuje karcinom ze sběrných kanálků (tzv. onkocytom) a medulární karcinom (tzv. Ductus Bellini karcinom) [2]. Základním prognostickým ukazatelem stále zůstává rozsah onemocnění definovaný TNM klasifikací. V případě karcinomů omezených pouze na vlastní ledvinu, nepřesahujících 7cm, dosahuje pětiletého přežití až 90% pacientů, u větších nádorů cca 60% pacientů, při postižení uzlin či invaze do okolních velkých žil maximálně 40% a při metastatickém rozsevu přibližně 5% pacientů [3–4]. K vyhledání pacientů se špatnou prognózou lze rovněž použít několik kombinovaných klinickopatologických skórovacích systémů, např. Memorial Sloan-Kettering Cancer Center kritéria (www.mskcc.org) nebo nomogramy University of Montreal (www.umontreal.ca).
Základní léčebnou modalitou je chirurgie, která jako jediná má kurativní potenciál, v případě inoperabilního a diseminovaného onemocnění na významu nabývají systémová imunoterapie, cytostatická léčba a radioterapie. Renální karcinom je nicméně vysoce radio - a chemorezistentní onemocnění a podobně i v případě imunoterapie nepřekračuje počet léčebných odpovědí 20%. Z tohoto důvodu není adjuvantní léčba dosud standardním postupem [3–4]. V paliativním přístupu se až do současnosti uplatňovala pouze imunoterapie založená na aplikaci interferonu alfa a/nebo interleukinu-2. V posledních letech došlo k výraznému rozšíření terapeutických možností díky rozvoji cílené terapie působící na úrovni signálních drah angiogeneze, buněčné proliferace a apoptózy. V klinické praxi jsou v současnosti používány především nízkomolekulární inhibitory tyrozinkináz sunitinib, sorafenib a temsirolimus a monoklonální protilátka bevacizumab zaměřená proti vaskulárnímu endoteliálnímu faktoru (VEGF) [5–7]. Do popředí zájmu se dostávají také moderní imunoterapeutické postupy založené na aktivním ovlivňování imunitního systému, především cestou protinádorových vakcín na bázi dendritických buněk [8]. Vývoj většiny nových preparátů určených pro tzv. cílenou terapii (targeted therapy) by ovšem nebyl možný bez současného rozvoje genomických metod umožňujících komplexní pohled na molekulární podstatu karcinogeneze. Moderní genomické přístupy založené na mikročipových technologiích byly v posledních letech opakovaně využity rovněž ve výzkumu renálního karcinomu. Publikované práce popisují schopnost mikročipů odlišit nádorovou tkáň od normálního renálního parenchymu, klasifikovat zhoubné novotvary ledviny podle histologických podtypů, identifikovat genové profily predikující metastazování renálního karcinomu a determinující prognózu jednotlivých pacientů [9–10]. Cílem tohoto přehledu je shrnout výsledky z dosud provedených mikročipových studií u renálního karcinomu a prezentovat jejich potenciální uplatnitelnost v diagnostických a léčebných postupech.
DNA a mikroRNA čipové technologie
Technologie DNA čipů (DNA microarrays) umožňuje ve velmi krátkém čase paralelně monitorovat expresi tisíců genů, případně celý lidský genom na úrovni RNA (tedy transkriptom) [11–13]. DNA čipy lze definovat jako miniaturizované zařízení nesoucí na svém povrchu imobilizované fragmenty nukleových kyselin, označované jako sondy, v přesně daném uspořádání. Tyto fragmenty jsou sekvenčně odvozeny tak, aby mohly specificky hybridizovat s volnými molekulami nukleových kyselin izolovanými z analyzovaného vzorku, který je předem fluorescenčně označen za účelem posthybridizační detekce [11]. Signál generovaný ohraničeným polem odpovídajícím dané sondě pak odráží hladinu exprese mRNA daného genu v analyzovaném vzorku. Po detekci, denzitometrické kvantifikaci a normalizaci intenzit signálu pomocí specializovaného softwaru je vytvořen tzv. profil genové exprese analyzovaného vzorku, který je pak možné srovnávat s expresními profily dalších vzorků. Díky výrazným alteracím v expresních profilech nádorové buňky je ideální aplikací pro tuto technologii právě onkologický výzkum. Klíčovou studii z hlediska onkologické prognostiky provedli van’t Veerová et al [14], kteří analyzovali profily genové exprese primárních nádorů pacientek s karcinomem prsu a pomocí exprese 70 genů byli schopni predikovat vznik metastáz s 89% přesností [14]. Ve srovnání s dosud klinicky používanými prognostickými ukazateli se jedná o velmi přesný odhad relapsu onemocnění, který, pokud by byl potvrzen, by znamenal možnost výraznější individualizace protinádorové léčby. Řada pacientek s karcinomem omezeným pouze na oblast prsu a s nízkým rizikem relapsu onemocnění by tak nemusela vůbec podstupovat adjuvantní chemoterapii. K validaci prediktivního potenciálu zmíněného 70genového profilu byla otevřena klinická studie MINDACT (Microarray In Node negative Disease may Avoid ChemoTherapy). Probíhá v síti 40 výzkumných pracovišť z 21 zemí s celkovým rozpočtem přibližně 20 mil. eur a očekává se, že do ní bude zařazeno přibližně 5 000 pacientek [15]. K získání dalších informací o použití DNA čipů v oblasti onkologického výzkumu odkazujeme čtenáře na již publikované přehledové práce [12–13].
Nejnovějším z molekulárněbiologických přístupů charakterizace nádorů je jejich studium pomocí profilů exprese mikroRNA (miRNA). MiRNA jsou poslední z dosud objevených ribonukleových kyselin (RNA). Jedná se o nekódující jednořetězcové RNA o délce 18–25 nukleotidů, jejichž hlavní funkcí je regulace genové exprese na úrovni mediátorové RNA (mRNA) [16]. V závislosti na míře komplementarity s cílovou mRNA využívají miRNA dva rozdílné mechanizmy posttranskripční regulace genové exprese, které vedou buď přímo k degradaci cílové mRNA, nebo k represi její translace a následně k poklesu hladin jejího proteinového produktu. Dle bioinformatických modelů jsou miRNA schopny regulovat až jednu třetinu lidských genů, včetně významných onkogenů či nádorových supresorů. Geny regulované pomocí miRNA se uplatňují jak v iniciálních fázích procesu karcinogeneze, tak i v regulaci nádorové invazivity a metastazování. Kromě toho se účastní i řady dalších procesů, např. lékové rezistence [17–19]. Zajímavá je rovněž skutečnost, že přibližně 50% miRNA genů se nachází na fragilních částech chromozomů, jejichž ztráta nebo amplifikace je často detekována u nádorových onemocnění. Z tohoto pohledu jsou i nekódující miRNA nositelkami důležité genetické informace, jejíž regulace je narušena, nebo dochází k její ztrátě v průběhu nádorové transformace. Analýza expresních profilů miRNA je proto stále častěji využívána pro účely molekulární charakterizace nádorových onemocnění, analogicky se studiemi založenými na DNA čipech a profilování kódujících transkriptů.
Expresní čipové technologie a renální karcinom
V posledních osmi letech bylo publikováno přes dvacet studií, ve kterých bylo ve výzkumu renálního karcinomu využito DNA čipových technologií. Tyto studie byly většinou zaměřeny na charakterizaci profilu genové exprese renálního karcinomu a jejich výstupem byla většinou sada genů, jejichž exprese se významně lišila mezi nádorovou tkání a normálním parenchymem ledviny. Tímto způsobem byly identifikovány stovky nových genů, potenciálních markerů renální karcinogeneze. Bohužel mikročipové analýzy je vždy nutné podrobovat kritickému pohledu, neboť jak je při podobných experimentech obvyklé, sady genů nalezených jednotlivými autory se vzájemně značně liší. Tato skutečnost může být způsobena zejména nízkým počtem analyzovaných vzorků, nedostatečně homogenními soubory pacientů, rozdílnými limity pro kvalitu izolované RNA, použitím různých čipových platforem (cDNA čipy – Agilent, oligonukleotidové čipy – Affymetrix, kuličkové čipy – Illumina, Real-Time PCR čipy – Applied Biosystems) a stejně tak variabilitou statistických metod a různými kritérii statistické významnosti [20–22].
Přes všechny tyto limitace existují geny, které byly opakovaně identifikovány ve více studiích. Příklady genů nadměrně exprimovaných v renální nádorové tkáni jsou VEFG, endotelin 1, glukózový přenašeč GLUT9, IGF vázající protein 3 v nádorech typu cRCC, alfa metyl-CoA racemáza u pRCC nebo KIT (CD117) u chrRCC [10]. Tyto geny se podílejí zejména na regulaci buněčného metabolizmu, angiogeneze a signální transdukce, a rozšiřují tak spektrum již dříve popsaných genetických abnormalit u RCC (např. mutace genu von Hippel-Lindau, onkogenu MET a receptoru pro epidermální růstový faktor /EGFR/) [23–24]. Podobně jako v případě karcinomu prsu uvedeném výše byly i u RCC zjištěny specifické skupiny genů (genové profily), jejichž vzájemná exprese je spojena s prognózou onemocnění. Tyto profily mají potenciál pro užití v diagnostice a léčbě pacientů s RCC. Za tímto účelem však bude nutné provést jejich validace formou klinických studií.
Velice slibné jsou výsledky prací zaměřených na profilování exprese mikroRNA u pacientů s RCC. Zatímco k jednoznačnému odlišení renálního karcinomu od renální nenádorové tkáně jsou zapotřebí skupiny čítající desítky genů (mRNA), na úrovni miRNA je tento počet významně nižší, neboť jedna miRNA může regulatovat desítky až stovky cílových mRNA. To dokazuje i práce německých autorů, kteří na základě kombinace pouze dvou miRNA, miR-141 a miR-155, byli schopni správně identifikovat 97% testovaných vzorků RCC mezi vzorky normální renální tkáně [25].
Profily genové exprese renálního karcinomu a nenádorové tkáně: Lze diagnostikovat renální karcinom pomocí čipové technologie?
Technologie DNA čipů byla nejčastěji použita ke srovnávání profilů genové exprese cRCC a nenádorové renální tkáně (NRT). Cílem těchto studií je bezchybně identifikovat renální karcinom na molekulární úrovni a zároveň rozšířit naše znalosti o molekulárněbiologické povaze tohoto klinicky heterogenního nádorového onemocnění. Dosud nalezené geny je možné rozdělit do skupin na základě jejich biologické funkce. Příkladem je práce Boerové et al [26], kteří použili cDNA čipy k analýze biologického materiálu z 32 primárních tumorů a 5 metastáz RCC a identifikovali 1 738 genů rozdílně exprimovaných ve srovnání s nenádorovou tkání parenchymu ledviny. Geny zapojené do buněčné adheze, metabolizmu nukleových kyselin a nukleotidů byly u RCC zvýšeně exprimovány, zatímco hladiny genů spojené s iontovou homeostázou, transportem elektronů a metabolizmem kyslíku vykazovaly sníženou expresi. S podobným designem bylo provedeno několik dalších studií [27–32]. Na souboru 18 RCC a jejich paralelní nenádorové tkáni provedli analýzu expresních profilů pomocí čipů Affymetrix Lenburg et al [33] a identifikovali 1 234 rozdílně exprimovaných genů. Svoje pozorování rozšířili o metaanalýzu publikovaných studií, ve které zjistili, že 800 jimi identifikovaných genů nebylo nalezeno v žádné z předchozích studií, kdežto pouze 37 genů bylo již dříve identifikováno minimálně ve třech studiích. Tyto geny byly spojeny s biologickými procesy, jako je hypoxie, angiogeneze, apoptóza, odpověď na interferon a léková rezistence [33]. V podobné studii Jonese et al [34] bylo mezi vzorky 32 cRCC a 23 normálních parenchymů ledviny nalezeno 1 359 genů se zvýšenou a 493 se sníženou expresí u RCC. Stejně tak i Jones byl schopen rozdělit identifikované geny do několika hlavních skupin na základě jejich biologické funkce: angiogeneze (např. VEGF, angiopoietin like 4 a 2, bFGF, ECGF1, endotelin 1, TGF beta 1 a 2), invazivita/migrace/adheze (např. MMP14, CXCR1, CXCR4), tyrozinkinázové receptory (např. FLT1, AXL, EGFR). Mezi touto genovou sadou a sadami publikovanými Lenburgem et al [33] existoval významný překryv, přestože byly použity rozdílné DNA čipové platformy a rozdílné bioinformatické algoritmy. Stejný tým studoval také různé podtypy RCC (pRCC, chrRCC a onkocytomy) a nádory ledvinné pánvičky, přičemž profily genové exprese rozdělily tyto nádory do skupin v souladu s jejich histopatologickými fenotypy [33]. Překvapivě existuje sada 31 genů, která vykazuje zvýšenou expresi napříč všemi podtypy RCC i jinými národy ledvin. Tato sada genů v sobě zahrnuje geny se známou funkcí v nádorové biologii: transformující růstový faktor β2 (TGF β2), disintegrin a metaloproteináza 12 (MMP12) a proteinkináza Cδ, naznačující význam těchto běžných biologických mechanizmů napříč všemi typy renálních nádorů. Deregulace signální dráhy TGF β byla již dříve asociována s kancerogenezí a progresí RCC [35]. Disintegrin a MMP12 byly zase úspěšně studovány v souvislosti s prognózou RCC [36]. V případě mikroRNA bylo provedeno rovněž několik studií zaměřených na porovnání exprese miRNA mezi renálními karcinomy a normálním parenchymem ledviny. Gottardo et al [37] identifikovali sadu čtyř miRNA se signifikantně zvýšenou expresí (miR-28, miR-185, miR-27, and let-7f-2) v tkáni renálního karcinomu oproti kontrolním vzorkům tkáně zdravé ledviny. V další práci používající miRNA čipy nesoucí sondy pro 470 miRNA, Nakada et al [38] pozorovali u RCC snížené hladiny miR-141 a miR-200c, které validovali metodou Real-Time PCR, a jako cíle regulované těmito miRNA potvrdili ZFHX1B a E-kadherin. Jung et al [25] na souboru 12 párových biopsií cRCC a normální renální tkáně stanovili specifický miRNA profil cRCC, charakterizovaný zvýšenou expresí miR-16, miR-452*, miR-224, miR-155 a miR-210 a snížením exprese miR-200b, miR-363, miR-429, miR-200c, miR-514 miR-141. Změny hladin exprese uvedených miRNA byly validovány metodou Real-Time PCR na souboru 72 párových biopsií cRCC a normální renální tkáně. Na tomto testovacím souboru bylo možné odlišit cRCC od zdravé tkáně pouze na základě exprese miR-141 miR-155, a to s 98% přesností [25]. V práci kanadského týmu se podařilo u cRCC identifikovat 33 deregulovaných miRNA, z nichž 21 bylo zvýšeno (např. miR-21, miR-210, miR-106a, miR-106b, miR-15a) a 12 sníženo (např. miR-200c, miR-200b, miR-720, miR-150) ve srovnání se zdravou tkání [39]. Napříč všemi prezentovanými studiemi bylo pozorováno snížení exprese miR-200c, velice frekventně také snížení exprese miR-141, a naopak nárůst exprese miR-210. Všechna výše uvedená pozorování potvrzují, že miRNA hrají významnou roli v kancerogenezi RCC a je třeba je brát v úvahu v rámci komplexního vnímání molekulární patologie tohoto onemocnění.
Molekulární klasifikace renálního karcinomu: Lze pomocí mikročipů určit, o jaký podtyp renálního karcinomu se jedná?
Diagnostika a klasifikace nádorových onemocnění na molekulární úrovni je v současnosti nezbytným předpokladem ke správné léčbě již u řady diagnóz (např. karcinom prsu, kolorektální karcinom, difuzní velkobuněčný lymfom). V případě renálního karcinomu existuje několik histologických podjednotek (podrobně viz úvod), ale podobně jako u jiných nádorů se ukazuje, že i přes stejný morfologický základ (v daném případě renální tubulární epitel) dochází u jednotlivých podjednotek v průběhu karcinogeneze k řadě odlišných genetických alterací.
Pomocí DNA čipů bylo zjištěno, že morfologická diverzita RCC má významné koreláty v profilech genové exprese. Např. Schuetz et al [30] popsali specifické expresní profily ve vztahu k jednotlivým histologickým podtypům RCC. Jak světlobuněčný (cRCC), tak papilární (pRCC) renální karcinom vykazovaly profil genové exprese charakteristický pro proximální nefron, zatímco u chromofobního renálního karcinomu (chrRCC) a onkocytomu byly nadměrně exprimovány především geny typické pro distální nefron [30]. Sultmann et al [40] jednoznačně prokázali rozdílnost expresních profilů cRCC a chrRCC ve studii s 87 RCC a 25 vzorky normálního renálního parenchymu. Ve své práci navíc použili DNA čipy specificky konstruované pro výzkum zhoubných nádorů ledvin. Tyto čipy nesly na svém povrchu sondy 4 207 genů asociovaných s renální tkání. Jones et al [34] stanovili pro každý z pěti studovaných histologických podtypů RCC genový profil čítající 30 genů. Použitím celé sestavy 150 genů byli následně schopni zpětně identifikovat jednotlivé podtypy RCC se 100% přesností. Tato skupina genů, jejichž seznam je součástí oddílu „Supplementary data“ autorovy práce (http://www.bidmegenomics.org/KidneyCancer/index.html), byla úspěšně validována nezávislou laboratoří na rozdílném souboru pacientů při použití odlišné čipové technologie [40]. Rozdílnost a průniky expresních profilů jednotlivých histologických podtypů RCC demonstruje množinový diagram (obr. 1) vytvořený na základě dat získaných Jonesem et al [34]. Rovněž na úrovni miRNA byly identifikovány expresní profily specifické pro jednotlivé histologické podtypy RCC [41]. Konkrétně exprese miR-423 a miR-203 byla signifikantně zvýšená u cRCC ve srovnání s pRCC. Naproti tomu u onkocytomu byla miR-203 snížená ve srovnání s chrRCC [41].
Obr. 1. Množinový diagram znázorňující souvislost jednotlivých histologických podtypů Rcc (cRCC – světlobuněčný renální karcinom, pRCC – papilární renální karcinom, chrRCC – chromofobní renální karcinom) na úrovni profi lů genové exprese. Čísla udávají počty genů se signifi kantně rozdílnou expresí. Upraveno podle Jonese et al [9]. ![Množinový diagram znázorňující souvislost jednotlivých histologických podtypů Rcc (cRCC – světlobuněčný renální karcinom, pRCC – papilární renální karcinom, chrRCC – chromofobní renální karcinom) na úrovni profi lů genové exprese. Čísla udávají počty genů se signifi kantně rozdílnou expresí. Upraveno podle Jonese et al [9].](https://www.prelekara.sk/media/cache/resolve/media_object_image_small/media/image/b71ce2d06cd7ca3ab568f3805f4dd8c1.jpg)
V případě renálního karcinomu je pozoruhodné, že přes klinicky heterogenní onemocnění, a to i uvnitř jednotlivých histologických podjednotek, profilování genové exprese nepřineslo žádné změny v jeho klasifikaci. To je v rozporu s očekáváním odborné veřejnosti, jež předpokládala změny podobné těm, ke kterým došlo např. u difuzního velkobuněčného lymfomu nebo u karcinomu prsu. Z tohoto pohledu molekulární klasifikace RCC kopíruje histologické podtypy RCC. Identifikované genové skupiny ovšem umožní hlubší pochopení molekulární povahy jednotlivých histologických podtypů RCC.
Využití DNA a mikroRNA čipů ke stanovení prognózy pacientů s renálním karcinomem: Jaké je riziko metastazování?
Ve své podstatě prvním a dosud jediným v klinické praxi široce využívaným prognostickým systémem renálního karcinomu je TNM klasifikace. Přestože se přežití pacientů s renálním karcinomem jednoznačně odvíjí od klinického stadia choroby i uvnitř jednotlivých skupin (I. až IV. klinické stadium), se může dosažená délka času do progrese choroby (disease-free survival – DFS) a celkového přežití (overall survival – OS) mezi jednotlivými pacienty významně lišit. Mikročipové technologie nabízejí atraktivní nástroj k hledání rozdílů v genové expresi karcinomů ledviny mezi pacienty s výrazně odlišnými časy DFS a OS.
První studie využívající genomický přístup ke klasifikaci RCC podle rozdílné prognózy byla provedena na souboru 29 pacientů s cRCC pomocí cDNA čipů nesoucích 21 632 sond [42]. Takahashi et al [42] identifikovali soubor 40 genů umožňující rozdělit pacienty s cRCC do skupiny s dobrou a špatnou prognózou. Mezi těmito geny se objevily i některé již dříve identifikované, jako např. TGF β, TGF βRII nebo TIMP3. Je zajímavé, že u pacientů se špatnou prognózou byla pozorována snížená exprese TGF βRII a TGF βRIII a dalších genů spojených s TGF β signální dráhou, zatímco samotné TGF β2 se v těchto cRCC vyskytovalo ve zvýšených hladinách [42]. Vasselli et al [43] pomocí DNA čipů na souboru 58 pacientů s metastazovaným RCC identifikovali 45 genů, na základě jejichž vzájemné exprese mohli být pacienti stratifikováni do skupiny s nízkým nebo vysokým rizikem časného úmrtí v důsledku progrese renálního karcinomu. U pacientů s nízkým rizikem byl dosažen medián OS 556 dnů (tříleté OS téměř 40% případů), u pacientů s vysokým rizikem 180 dnů (tříleté přežití méně než 10% případů (p < 0,001)). Nejvýznamnějším molekulárním prognostickým ukazatelem z uvedených 45 genů byl gen kódující VCAM1 (vaskulární adhezivní molekula VCAM1) [43]. Další gen identifikovaný pomocí DNA čipů, jehož význam v prognóze cRCC je v současnosti studován, je protein spojený s diferenciací tukové tkáně (ADFP). Kromě adipocytů se ADFP zvýšeně vyskytuje i v řadě jiných tkání, např. v buňkách mamárních duktů, v Sertoliho a Leydigových buňkách. ADFP patří mezi geny indukované hypoxií, přičemž hlavním transkripčním faktorem, který zprostředkovává tuto odpověď, je HIF (hypoxia-inducible factor) [44–45]. Yao et al [44] nejdříve provedli analýzu profilu genové exprese u 33 vzorků RCC a 9 normálních vzorků ledviny, při které zjistili vyšší expresi ADFP u světlobuněčných renálních karcinomů a následně na konsekutivním souboru 423 pacientů se sporadickým cRCC potvrdili pozitivní prediktivní význam ADFP [45]. Exprese ADFP byla výrazně vyšší u pacientů bez přítomnosti systémových klinických symptomů nádorového onemocnění, u karcinomů dobře diferencovaných, lokálně méně pokročilých. Naopak nižší expresi ADFP zaznamenali u špatně diferencovaných karcinomů nebo pacientů s již metastatickým onemocněním. Hladina ADFP významně korelovala s DFS i OS [45].
Možnost přesně predikovat metastatický potenciál renálního karcinomu již v době diagnózy by mohla významně ovlivnit léčebný plán. Kosari et al [46] rozdělili svůj soubor pacientů s cRCC na část s agresivním průběhem onemocnění, která podlehla nemoci do čtyř let od nefrektomie, a na část s relativně příznivým průběhem choroby. Pomocí DNA čipů provedli analýzu profilů genové exprese v obou souborech a nalezli sadu 35 genů, jejichž exprese se mezi oběma soubory statisticky významně lišila. Své výsledky verifikovali na nezávislém souboru pacientů metodou Real-Time PCR a mezi analyzovanými geny identifikovali anti apoptotický survivin, jehož vysoké hladiny byly spojeny s kratším přežitím pacientů s cRCC. Souvislost survivinu a přežívání pacientů s cRCC ověřil tento tým rovněž imunohistochemicky na proteinové úrovni [46].
Zatímco profily genové exprese specifické pro jednotlivá klinická stadia cRCC nebyly dosud použitím DNA čipů nalezeny, další prognostický genový profil obsahující 155 genů stanovili Jones et al na základě srovnání profilů genové exprese vzdálených metastáz cRCC a primárních tumorů klasifikovaných v TNM klasifikaci T1 (tumor nepřesahující 7cm, omezený pouze na samotnou ledvinu) [9,34,47]. Expresi uvedených 155 genů následně vyšetřili ve vzorcích primárních cRCC o rozsahu T3 (nádor se šíří do velkých vén nebo postihuje přímo nadledvinu nebo perirenální tkáň, ale ne přes Gerotovu fascii). Výsledek genové exprese rozdělil T3 tumory na dvě skupiny. Skupina T3 tumorů, která měla profil genové exprese 155 genů podobný profilu vzdálených metastáz, měla v době chirurgického výkonu vzdálené metastázy skutečně přítomny. Druhá skupina T3 tumorů s profilem genové exprese podobným T1 tumorům v době chirurgického výkonu vzdálené metastázy neměla (obr. 2). Z dendrogramu na obr. 2 je patrné, že primární nádory o stejném rozsahu (T3) mohou mít rozdílné biologické vlastnosti, které determinují vznik vzdálených metastáz. Tyto vlastnosti přitom nemohou být zjištěny při rutinním histologickém vyšetření.
Obr. 2. Část 155genového metastatického profilu cRCC , vytvořeného na základě srovnání expresních profilů vzdálených metastáz cRCC a primárních tumorů o rozsahu T1 (dle TNM klasifikace). Tento profil následně aplikovaný na cRCC o rozsahu T3 rozdělil T3 tumory na dvě skupiny. Skupina T3 tumorů, která měla profil genové exprese 155 genů podobný profilu vzdálených metastáz, měla v době chirurgického výkonu vzdálené metastázy skutečně přítomny. U pacientů s T3 tumory s profilem genové exprese podobným T1 tumorům v době chirurgického výkonu vzdálené metastázy přitomny nebyly. 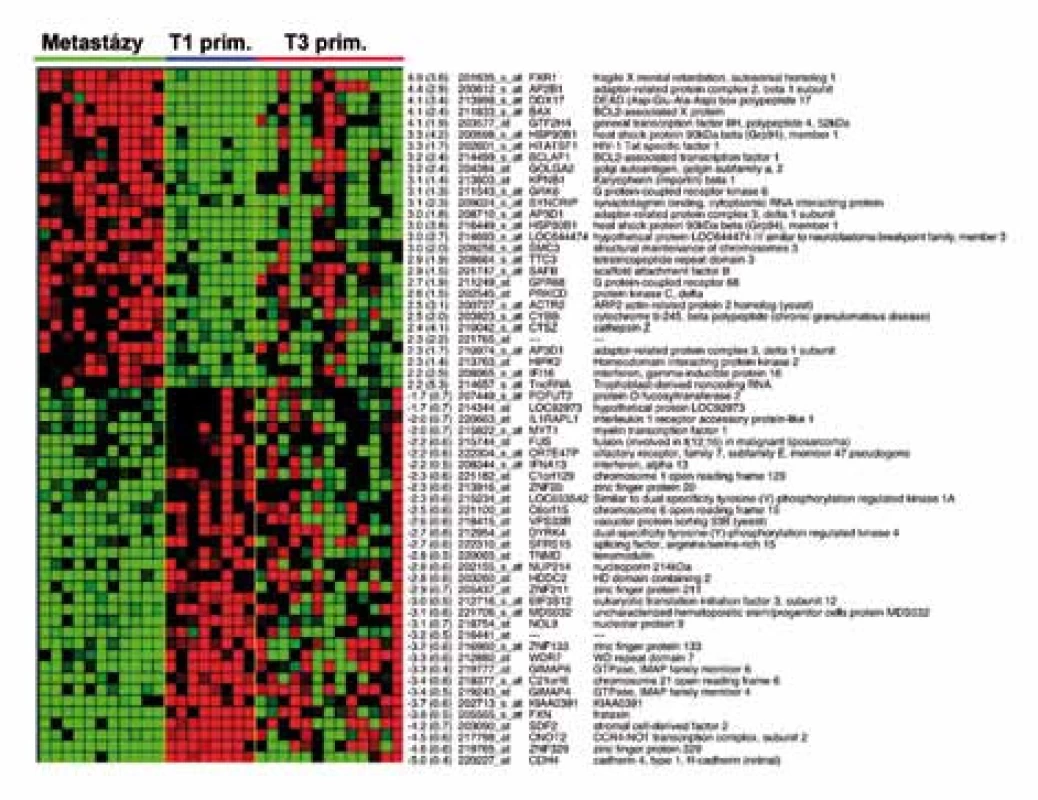
Identifikovaný 155genový profil zahrnoval řadu genů s již popsaným významem v kancerogenezi (např. proteinkináza Cδ, transkripční faktor POU5F1/OCT4, heat shock protein 90beta, kaveolin 1, annexin A4, katepsin Z. Jones na svém souboru vyzkoušel aplikovat i 128genový profil, objevený bostonskými autory na modelu karcinomů jiného než renálního původu, který predikuje vznik metastáz obecně u epiteliálních nádorů [48]. S použitím tohoto „obecného profilu metastazování“ dosáhl správného rozdělení více než 80% primárních tumorů do dvou výše definovaných skupin. Jak přesně interpretovat, že dva odlišné genové profily shodně seskupily světlobuněčné renální karcinomy na základě jejich schopnosti zakládat metastázy, není jasné. Rozhodně však tato skutečnost potvrzuje biologickou odlišnost primárních tumorů schopných časně metastazovat od nádorů méně agresivních. Tuto odlišnost lze přitom stanovit pomocí profilu genové exprese. Z klinického pohledu je pak možné včas rozpoznat pacienty s vysokým rizikem vzniku metastáz a zlepšit jejich prognózu individualizací jejich sledování a včasným zahájením správné léčby.
Na úrovni miRNA definovali odlišné prognostické skupiny pacientů (ve vztahu k pětiletému přežití) Petillo et al (obr. 3) [41]. Nádory se špatnou prognózou byly charakterizovány především zvýšenou expresí pre miR-32, což je prekurzor, ze kterého je aktivní miR-32 tvořena účinkem ribonukleázy Dicer. Studie zaměřené na zdokonalení stagingu cRCC na základě expresních profilů miRNA probíhají také na Klinice komplexní onkologické péče Masarykova onkologického ústavu v Brně.
Obr. 3. Skupina miRNA umožňující rozdělit pacienty s cRCC na skupiny s dobrou a špatnou prognózou. Červená a modrá barva znamená zvýšenou, resp. sníženou expresi dané miRNA ve srovnání s nenádorovou renální tkání. Předpona S znamená prekurzorovou formu dané miRNA. Upraveno podle Petilla et al [41]. ![Skupina miRNA umožňující rozdělit pacienty s cRCC na skupiny s dobrou a špatnou prognózou. Červená a modrá barva znamená zvýšenou, resp. sníženou expresi dané miRNA ve srovnání s nenádorovou renální tkání. Předpona S znamená prekurzorovou formu dané miRNA. Upraveno podle Petilla et al [41].](https://www.prelekara.sk/media/cache/resolve/media_object_image_small/media/image/ceec3363098b0b86a5ffd81272cae4ae.jpg)
Závěr: vize klinického využití moderních genomických metod u pacientů s renálním karcinomem
DNA i mikroRNA čipové technologie byly již úspěšně implementovány do výzkumu nádorových onemocnění. V této oblasti bylo největšího pokroku dosaženo u nehodgkinských lymfomů a karcinomu prsu, kde DNA čipy významnou měrou přispěly k vytvoření molekulárních klasifikací uvedených chorob, jež mají jednoznačný prognostický potenciál a dopad na léčbu pacientů. V současné době u karcinomu prsu probíhají klinické studie s použitím DNA čipů, čítající tisíce pacientek. Zda nabudou čipové technologie podobného významu také u renálního karcinomu, je předčasné odhadovat, neboť dosud nebylo provedeno tolik studií jako v případě dvou výše uvedených malignit. Nicméně dosažené výsledky jsou velice povzbudivé. Pomocí DNA a mikroRNA čipů byly identifikovány jednotlivé geny a miRNA mající prognostický význam u cRCC. Obě čipové platformy lze použít k diferenciaci mezi nádorovou tkání a normálním renálním parenchymem, v případě nádoru lze rozlišit jednotlivé podtypy RCC. Čipové technologie si nekladou za cíl nahradit histologické vyšetření bioptované tkáně, navíc tkáň vyšetřovaná pomocí čipů musí být z řady důvodů zpracována a zhodnocena patologem. Lze ovšem předpokládat, že do budoucna budou aplikována obě vyšetření paralelně, přičemž čipové technologie významnou měrou doplní informace získané z rutinního histologického vyšetření. Tyto informace budou základem molekulárního stagingu a molekulární klasifikace nádorových onemocnění, s přímým dopadem do klinické praxe.
Práce byla podpořena grantem Ministerstva zdravotnictví ČR IGA NS 10361-3/2009, projektem MZ0MOU2005 a grantem MŠMT NPV II – 2B 06058 LF 2921.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
The authors declare they have no potential conflicts of interest concerning drugs, pruducts, or services used in the study.Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
The Editorial Board declares that the manuscript met the ICMJE “uniform requirements” for biomedical papers.RNDr. Ondřej Slabý, Ph.D.
Klinika komplexní onkologické péče
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: slaby@mou.cz
Zdroje
1. Dušek L, Mužík J, Kukásek M et al. Český národní webový portál epidemiologie nádorů [online]. Masarykova univerzita, 10. 1. 2008. http://www.svod.cz.
2. Yan BC, Mackinnon AC, Al-Ahmadie HA. Recent developments in the pathology of renal tumors: morphology and molecular characteristics of select entities. Arch Pathol Lab Med 2009; 133 : 1026–32.
3. Bukowski RM. Prognostic factors for survival in metastatic renal cell carcinoma: update 2008. Cancer 2009; 115 : 2273–2281.
4. Richie JP, Jonasch E, Kantoff PW. Renal Cell Carcinoma. In: Kufe WD, Bast RC, Hait WN et al. Holland-Frei Cancer Medicine, 7th ed. Hamilton (Canada): BC Decker 2006 : 1401–1410.
5. Motzer RJ, Hutson TE, Tomczak P et al. Overall survival and updated results for sunitinib compared with interferon alfa in patients with metastatic renal cell carcinoma. J Clin Oncol 2009; 27 : 3584–3590.
6. Escudier B, Eisen T, Stadler WM et al. Sorafenib for treatment of renal cell carcinoma: Final efficacy and safety results of the phase III treatment approaches in renal cancer global evaluation trial. J Clin Oncol 2009; 27 : 3312–3318.
7. Hudes G, Carducci M, Tomczak P et al. Temsirolimus, interferon alfa, or both for advanced renal-cell carcinoma. N Engl J Med 2007; 356 : 2271–2281.
8. Amato RJ. Vaccine therapy for renal cancer. Expert Rev Vaccines 2008; 7 : 925–935.
9. Jones J, Libermann TA. Genomics of renal cell cancer: the biology behind and the therapy ahead. Clin Cancer Res 2007; 13 : 685s–692s.
10. Jones J, Pantuck AJ. Genomics and proteomics in renal cell carcinoma: diagnosis, prognosis, and treatment selection. Curr Urol Rep 2008; 9 : 9–14.
11. Svoboda M, Michálek J. Úvod do technologie DNA čipů. Lék a Techn 2004; 35 : 67–75.
12. Perez-Diez A, Morgun A, Shulzhenko N. Microarrays for cancer diagnosis and classification. Adv Exp Med Biol 2007; 593 : 74–85.
13. Virtanen C, Woodgett J. Clinical uses of microarrays in cancer research. Methods Mol Med 2008; 141 : 87–113.
14. van't Veer LJ, Dai H, van de Vijver MJ et al. Gene expression profiling predicts clinical outcome of breast cancer. Nature 2002; 415 : 530–536.
15. Slabý O. Pokroky v čipových technologiích znamenají pokroky v biomedicínském výzkumu i klinické diagnostice. Klin Onkol 2007; 2 : 235–236.
16. Slaby O, Krekac D, Hrstka R et al. Zapojení mikroRNA do patogeneze nádorových onemocnění a možnosti jejich využití v diagnostické a prediktivní onkologii. Čas Lék Čes 2008; 147 : 25–31.
17. Garzon R, Calin GA, Croce CM. MicroRNAs in Cancer. Annu Rev Med 2009; 60 : 167–179.
18. Bartels CL, Tsongalis GJ. MicroRNAs: novel biomarkers for human cancer. Clin Chem 2009; 55 : 623–631.
19. Brown BD, Naldini L. Exploiting and antagonizing microRNA regulation for therapeutic and experimental applications. Nat Rev Genet 2009; 10 : 578–585.
20. Tibshirani R. A simple method for assessing sample sizes in microarray experiments. BMC Bioinformatics 2006; 7 : 106.
21. Hardiman G. Microarrays Technologies 2006: an overview. Pharmacogenomics 2006; 7 : 1153–8.
22. Tichý B, Svoboda M, Mayer J et al. Odběr a zpracování vzorků pro expresní DNA čipy. Klin Onkol 2006; 19 (Suppl 2): 350–354.
23. Lukešová Š, Kopecký O, Dvořák J et al. Význam genetických mutací a podstata poruchy angiogeneze u světlobuněčného karcinomu ledviny. Klin Onkol 2006; 19 : 290–292.
24. Křepelová A. Dědičné formy nádorů ledvin – spektrum predispozičních genů a jejich testování. Klin Onkol 2006; 19 (Suppl): 82–84.
25. Jung M, Mollenkopf HJ, Grimm C et al. MicroRNA profiling of clear cell renal cell cancer identifies a robust signature to define renal malignancy. J Cell Mol Med 2009. In press.
26. Boer JM, Huber WK, Sultmann H et al. Identification and classification of differentially expressed genes in renal cell carcinoma by expression profiling on a global human 31,500-element cDNA array. Genome Res 2001; 11 : 1861–1870.
27. Skubitz KM, Zimmermann W, Kammerer R et al. Differential gene expression identifies subgroups of renal cell carcinoma. J Lab Clin Med 2006; 147 : 250–267.
28. Liou LS, Shi T, Duan ZH et al. Microarray gene expression profiling and analysis in renal cell carcinoma. BMC Urol 2004; 4 : 9.
29. Higgins JP, Shinghal R, Gill H et al. Gene expression patterns in renal cell carcinoma assessed by complementary DNA microarray. Am J Pathol 2003; 162 : 925–932.
30. Schuetz AN, Yin Goen Q, Amin MB et al. Molecular classification of renal tumors by gene expression profiling. J Mol Diagn 2005; 7 : 206–218.
31. Gieseg MA, Cody T, Man MZ et al. Expression profiling of human renal carcinomas with functional taxonomic analysis. BMC Bioinformatics 2002; 3 : 26.
32. Diegmann J, Tomiuk S, Sanjmyatav J et al. Comparative transcriptional and functional profiling of clear cell and papillary renal cell carcinoma. Int J Mol Med 2006; 18 : 395-403.
33. Lenburg ME, Liou LS, Gerry NP et al. Previously unidentified changes in renal cell carcinoma gene expression identified by parametric analysis of microarray data. BMC Cancer 2003; 3 : 31.
34. Jones J, Otu H, Spentzos D et al. Gene signatures of progression and metastasis in renal cell cancer. Clin Cancer Res 2005; 11 : 5730–5739.
35. Copland JA, Luxon BA, Ajani L et al. Genomic profiling identifies alterations in TGFbeta signaling through loss of TGFbeta receptor expression in human renal cell carcinogenesis and progression. Oncogene 2003; 22 : 8053–8062.
36. Roemer A, Schwettmann L, Jung M et al. Increased mRNA expression of ADAMs in renal cell carcinoma and their association with clinical outcome. Oncol Rep 2004; 11 : 529–536.
37. Gottardo F, Liu CG, Ferracin M et al. Micro RNA profiling in kidney and bladder cancers. Urol Oncol 2007; 25 : 387–392.
38. Nakada C, Matsuura K, Tsukamoto Y et al. Genome-wide microRNA expression profiling in renal cell carcinoma: significant down-regulation of miR-141 and miR-200c. J Pathol 2008; 216 : 418–427.
39. Chow TF, Youssef YM, Lianidou E et al. Differential Expression Profiling of microRNAs and Their Potential Involvement in Renal Cell Carcinoma Pathogenesis. Clin Biochem 2009. In press.
40. Sultmann H, von Heydebreck A, Huber W et al. Gene expression in kidney cancer is associated with cytogenetic abnormalities, metastasis formation, and patient survival. Clin Cancer Res 2005; 11 : 646–655.
41. Petillo D, Kort EJ, Anema J et al. MicroRNA profiling of human kidney cancer subtypes. Int J Oncol 2009; 35 : 109–114.
42. Takahashi M, Rhodes DR, Furge KA et al. Gene expression profiling of clear cell renal cell carcinoma: gene identification and prognostic classification. Proc Natl Acad Sci U S A 2001; 98 : 9754–9759.
43. Vasselli JR, Shih JH, Iyengar SR et al. Predicting survival in patients with metastatic kidney cancer by gene-expression profiling in the primary tumor. Proc Natl Acad Sci U S A 2003; 100 : 6958–6963.
44. Yao M, Tabuchi H, Nagashima Y et al. Gene expression analysis of renal carcinoma: adipose differentiation related protein as a potential diagnostic and prognostic biomarker for clear-cell renal carcinoma. J Pathol 2005; 205 : 377–387.
45. Yao M, Huang Y, Shioi K et al. Expression of adipose differentiation related protein: a predictor of cancer-specific survival in clear cell renal carcinoma. Clin Cancer Res 2007; 13 : 152–160.
46. Kosari F, Parker AS, Kube DM et al. Clear cell renal cell carcinoma: gene expression analyses identify a potential signature for tumor aggressiveness. Clin Cancer Res 2005; 11 : 5128–5139.
47. Moch H, Artibani W, Delahunt B et al. Reassessing the Current UICC/AJCC TNM Staging for Renal Cell Carcinoma. Eur Urol 2009. In press.
48. Ramaswamy S, Ross KN, Lander ES et al. A molecular signature of metastasis in primary solid tumors. Nat Genet 2003; 33 : 49–54.
Štítky
Detská onkológia Chirurgia všeobecná Onkológia
Článek Neuroendokrinní nádory rekta
Článok vyšiel v časopiseKlinická onkologie
Najčítanejšie tento týždeň
2009 Číslo 5- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Kombinace metamizol/paracetamol v léčbě pooperační bolesti u zákroků v rámci jednodenní chirurgie
- Antidepresivní efekt kombinovaného analgetika tramadolu s paracetamolem
- Fixní kombinace paracetamol/kodein nabízí synergické analgetické účinky
-
Všetky články tohto čísla
- Neuroendokrinní nádory rekta
- DNA a mikroRNA čipové technologie v diagnostice a predikci u pacientů s renálním karcinomem
- Proteomická analýza nádorových buněk
- Studie evaporace vybraných cytostatik a propustnosti ochranných rukavic v rámci výzkumu profesní zátěže zdravotnických pracovníků exponovaných cytotoxickým protinádorovým léčivům (projekt CYTO)
- Polymorfizmus –2548 G/A v genu pro leptin u pacientek s endometriálním karcinomem
- Profily nízkomolekulárního proteomového spektra získané pomocí hmotnostní spektrometrie SELDI-TOF v sérech pacientů s diseminovaným maligním melanomem: pilotní studie
- Chronická gastrointestinální toxicita po zevní radioterapii karcinomu prostaty
- Úvaha nad vývojem screeningu kolorektálního karcinomu
- Pohled na vývoj onkologie ve 3. tisíciletí
- Zápis ze schůze výboru České onkologické společnosti dne 8. 9. 2009 v Liberci
- Klinická onkologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Neuroendokrinní nádory rekta
- Chronická gastrointestinální toxicita po zevní radioterapii karcinomu prostaty
- Studie evaporace vybraných cytostatik a propustnosti ochranných rukavic v rámci výzkumu profesní zátěže zdravotnických pracovníků exponovaných cytotoxickým protinádorovým léčivům (projekt CYTO)
- Proteomická analýza nádorových buněk
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



