-
Články
- Časopisy
- Kurzy
- Témy
- Kongresy
- Videa
- Podcasty
Molekulárně patologické profilování vybraných nádorů centrálního nervového systému metodou MLPA
Molecular pathological profiling of selected tumors of the central nervous system using the MLPA method
The current progress and increasing knowledge about the genetic causes of cancer opens up new possibilities for its treatment. However, it is necessary to combine the results obtained using classical pathological methods with sensitive, multiplex molecular pathological methods. The method that meets the required criteria is MLPA based on multiplex PCR reaction. This method detects both changes in gene copy number and DNA methylation and, last but not least, point mutations. The MLPA reaction is applicable to even highly fragmented DNA. At the same time, it is a robust method that can be performed on standard thermocyclers, the fluorescent tip label requires automatic sequencers. Up to 50 genetic markers can be tested in one reaction, a number that allows a diagnostic and prognostic conclusion. All these features lead to the routine use of MLPA analysis not only in diagnosis but also in cancer research. The present article aims to summarize the different types of MLPA reactions, its benefits, but also the potential pitfalls.
Keywords:
Molecular pathology – PCR – MLPA – Tumors of the central nervous system
Autori: Iva Dolinová 1; Eliška Tvrzníková 1; Veronika Janoušková 1; Aleš Vícha 2; Lenka Krsková 3; Tomáš Jirásek 4
Pôsobisko autorov: Oddělení molekulární genetiky a diagnostiky, Centrum PATOS, Krajská nemocnice Liberec, a. s. 1; Klinika dětské hematologie a onkologie 2. LF UK a Fakultní nemocnice Motol, Praha 2; Ústav patologie a molekulární medicíny 2. LF UK a Fakultní nemocnice Motol, Praha 3; Oddělení patologie, Centrum PATOS, Krajská nemocnice Liberec, a. s. 4
Vyšlo v časopise: Čes.-slov. Patol., 58, 2022, No. 3, p. 138-149
Kategória: Přehledový článek
Súhrn
Současný trend a zvyšující se znalosti o genetických příčinách vzniku a progresi zhoubných nádorů otevírá nové možnosti pro jejich léčbu. Nicméně je nutné výsledky získané pomocí klasických metod spojit s citlivými, molekulárně patologickými metodami. Metodou, která splňuje požadovaná kritéria, je multiplexní na ligaci závislá PCR reakce (MLPA) založená na multiplexní PCR reakci. Touto metodou jsou detekovány jak změny v počtu kopií genu, tak i metylační status DNA ve specifických oblastech a v neposlední řadě i bodové mutace. MLPA reakce je použitelná i na vysoce fragmentované DNA. Zároveň se jedná o metodu robustní, kterou lze provádět na standardních termocyklerech, fluorescenční značení vyžaduje analýzu pomocí automatického sekvenátoru. V jedné reakci lze testovat až 50 genetických markerů, což je počet, který umožňuje diagnostický a prognostický závěr. Všechny tyto vlastnosti vedou k rutinnímu využití MLPA analýzy nejen při diagnostice, ale i ve výzkumu nádorů. Předkládaný článek si klade za cíl popsat jednotlivé typy MLPA reakcí, její výhody, ale i potenciální úskalí.
Klíčová slova:
PCR – molekulární patologie – nádory centrálního nervového systému – MLPA
Metoda multiplexní na ligaci závislé PCR (MLPA) byla vyvinutá firmou MRC-Holland a poprvé popsána v roce 2002 (1). Původním záměrem metody bylo sledovat změny v počtu kopií segmentů DNA, takzvaných CNV Copy Number Variation (CNV), tedy delece nebo naopak duplikace v sekvenci DNA. Později MLPA analýza umožnila, za pomoci metylačně senzitivního restrikčního enzymu, charakterizovat míru metylace ve sledovaných oblastech. Pomocí MLPA reakce lze také detekovat vybrané bodové mutace.
Metoda MLPA je založena na principu PCR. Amplifikován ale není sledovaný úsek DNA, nýbrž sondy, které do cílových sekvencí hybridizují. Specifita reakce je zajištěna použitím dvou sond. V jedné reakci je možné analyzovat signál až 50 sond najednou. Sondy hybridizují do specifických oblastí genů rozmístěných po celém genomu, v závislosti na konkrétním panelu vyšetření. Jednotlivé sondy detekují oblasti 50–100 nukleotidů dlouhé, což umožňuje využití této metody i na vysoce fragmentovanou DNA. Zároveň tato vlastnost umožňuje detekci malých delecí, které mohou často zasahovat jednotlivé exony vyšetřovaných genů (1). Touto metodou lze tedy vyšetřit celé spektrum markerů, od genetických predispozic k dědičným onemocněním až po profilování nádorů. Firma MRC-Holland nabízí více než 400 panelů pro specifická vyšetření genetických změn.
Předkládaný článek vychází z review autorů Hömig-Hölzel a Savola z roku 2012 (2).
MLPA V PĚTI KROCÍCH
Průběh MLPA reakce lze rozdělit do pěti samostatných kroků. Prvním krokem je denaturace DNA při teplotě 98 °C, při níž dochází k rozvolnění vodíkových vazeb dvoušroubovice. Druhým krokem je hybridizace neboli navázání sond na specifické úseky genů. Přidávané sondy jsou složené z několika částí. Vždy obsahují univerzální PCR primer, jehož sekvence se nevyskytuje v lidském genomu. Druhou částí sondy je sekvence specifická ke sledovanému genu. Poslední částí sondy je „stuffer“ sekvence, jejíž délka je unikátní pro každou sondu. Tato sekvence slouží k finálnímu určení délky konkrétní sondy. V rámci jedné analýzy jsou sondy vždy navrženy tak, aby pokrývaly jak geny testovací, zodpovědné za onemocnění, tak i geny kontrolní, u nichž nedochází ke změně exprese. Do každé MLPA analýzy se, kromě testovaných, přidávají i kontrolní vzorky, u kterých nedochází ke změně počtu kopií genů. V případě bodových mutací musí být do každé reakce přidány i výrobcem připravené pozitivní vzorky. Samotný proces hybridizace trvá přibližně 16–20 hodin a probíhá při teplotě 60° (3). Třetím krokem je ligace spojující hybridizované sondy do jednoho fragmentu. Ligace probíhá při teplotě 54 °C a je zakončena inaktivací enzymu při 98 °C. Posledním krokem je PCR reakce, při které dochází k amplifikaci hybridizovaných sond pomocí univerzálních primerů (1,4,5). Vzniklé PCR amplikony jsou detekovány a kvantifikovány pomocí kapilární elektroforézy. Získaná data jsou následně hodnocena ve volně dostupném programu Coffalyser dodávaným firmou MRC-Holland (6). Při hodnocení výsledků dochází k porovnání intenzity signálu jak v rámci jedné analýzy (intra-vzorková normalizace dat), tak i mezi testovaným a kontrolním (referenčním) vzorkem (inter-vzorková normalizace dat). Stručné vyobrazení jednotlivých kroků je uvedeno v tabulce 1 a na obrázku 1.
Tab. 1. Jednotlivé kroky metody MLPA. 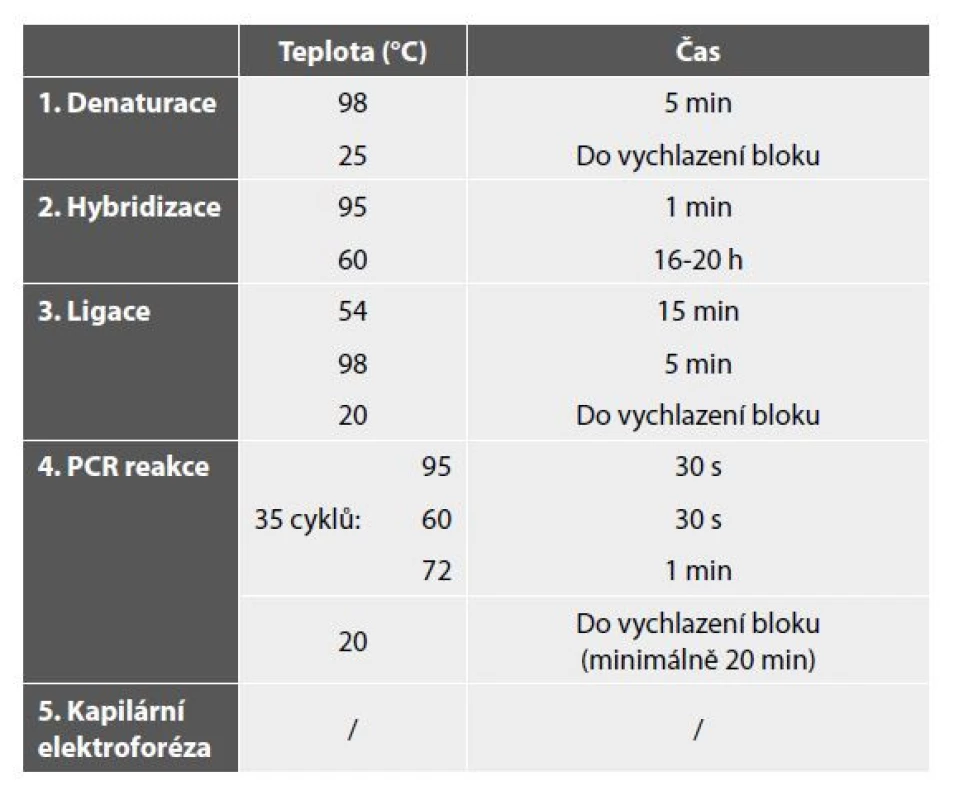
Obr. 1. Jednotlivé kroky metody MLPA (převzato ze 7). 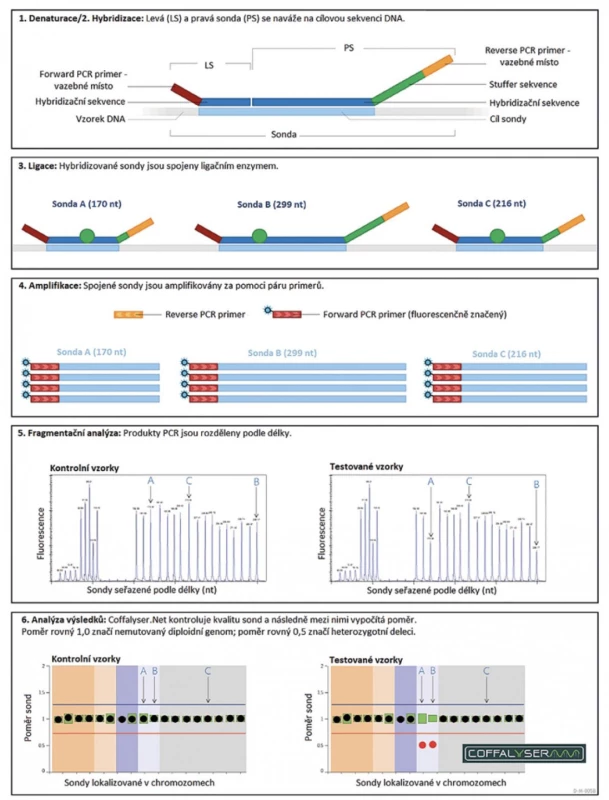
DETEKCE CNV (DUPLIKACE/DELECE) POMOCÍ MLPA ANALÝZY
Změna v počtu kopií sledovaného genu se projevuje snížením (delece) nebo zvýšením (amplifikace) intenzity signálu sledovaného genu. Vysoká reprodukovatelnost metody dovoluje rozlišovat mezi heterozygotní a homozygotní formou delece a zároveň dovoluje rozlišit mezi zmnožením a amplifikací. Při hodnocení delece/amplifikace konkrétního genu dochází k porovnání intenzity signálu u pacienta a zdravé kontroly. Jak již bylo stručně popsáno výše, dochází k porovnání signálu v rámci testovaného vzorku, ale i mezi vzorkem a referencí (kontrolním vzorkem beze změny v expresi sledovaných genů). S ohledem na charakter hodnocení nemohou být jako kontrolní sondy používány protoonkogeny či tumor supresorové geny, které jsou naopak spojovány s nádorovým bujením. Výběr referenčních sond je složitý proces, kdy jako zdrojová data slouží databáze jako například „Cosmic“ (8). Algoritmus hodnotící signály v programu Coffalyser rozděluje signály na testovací a referenční (podle typu sondy). Referenční signály slouží ke stanovení odezvy testovaného vzorku u nemutovaných genů. Tato odezva poté slouží k hodnocení jednotlivých testovacích signálů. Výsledkem jsou tedy jednotlivé poměry pro každou testovací sondu.
Těmito výpočty se zabraňuje hodnocení aberantních signálů a výdeji falešně pozitivních/negativních výsledků. Spolehlivá analýza dat je také podmíněna stabilní normalizační konstantou, jejíž výpočet vyžaduje shodu signálu referenčních sond mezi pacientem a kontrolním vzorkem ve více než 60 % (9). Vzhledem ke zvýšené genetické nestabilitě nádorových buněk je v MLPA směsích určených pro testování nádorových tkání použito nejméně 10 referenčních sond. Tyto sondy jsou rozmístěny na různé chromozomy, čímž dojde ke snížení rizika pro výpočet normalizační konstanty a zároveň ke zvýšení robustnosti metody. Zvýšení citlivosti při sledování změny exprese nádorových genů je naopak podpořeno umístěním více testovacích sond do cílového genu. Lze tak rozpoznat například deleci/amplifikaci pouze části genu. Výsledky analýzy MLPA poskytují informaci o situaci ve všech buňkách testovaného vzorku (nádorová tkáň). Pokud je procento přítomných nádorových buněk nízké, nelze genetické změny stanovit. Pro validní výsledky je nutné, aby bylo zastoupení nádorových buněk v testovaném vzorku minimálně 20 % (10). Podle našich zkušeností je však jejich optimální podíl ve vzorku 50 %. Při analýze nádorových tkání je velmi nutná součinnost molekulárně genetické a patologické laboratoře.
DETEKCE BODOVÝCH MUTACÍ POMOCÍ MLPA ANALÝZY
Obecným pravidlem MLPA reakce je nutnost úspěšné ligace hybridizovaných sond. Enzym Ligáza-65 používaný firmou MRC-Holland je vysoce citlivý na neshodu “mismatch“ v místě ligace a to zejména na 3´konci levé sondy. Tato vlastnost umožnila využití MLPA reakce pro detekci konkrétních bodových mutací (viz obr. 2)
Obr. 2. Detekce bodových mutací pomocí MLPA reakce (převzato z 2) 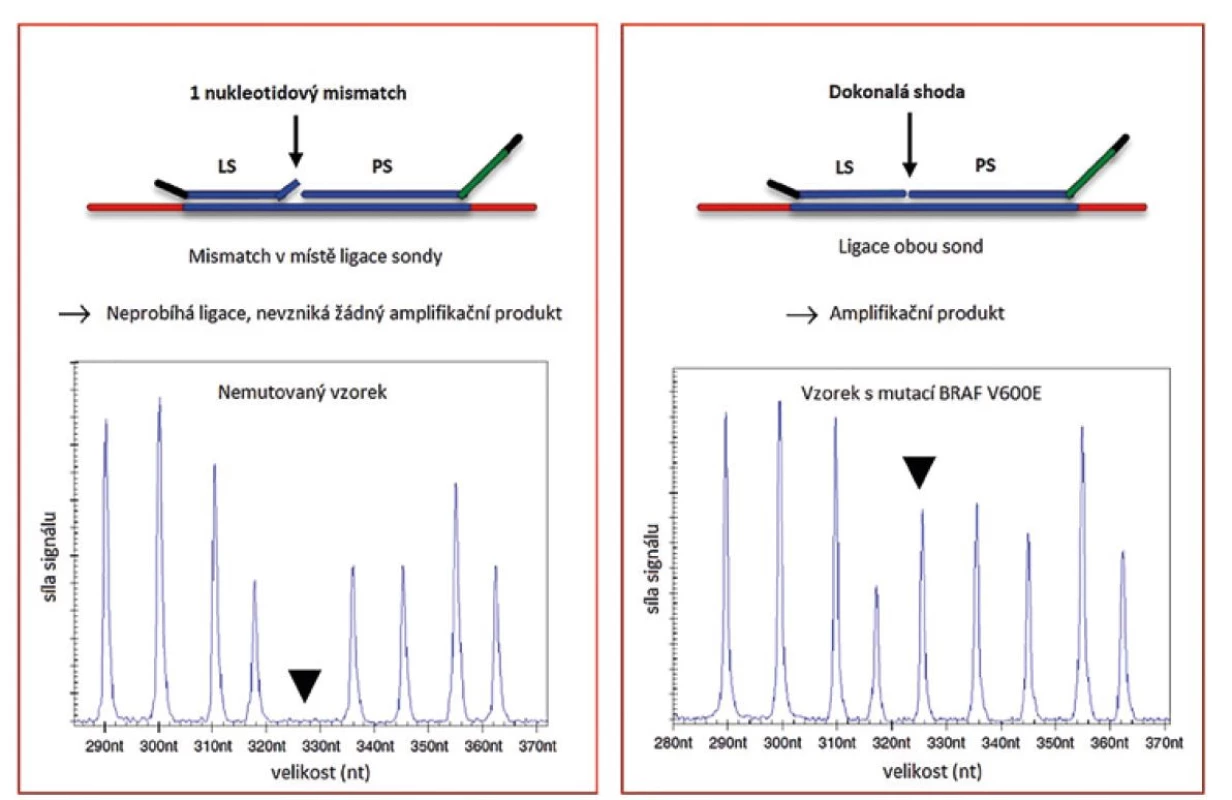
Mutačně specifické sondy jsou vždy navržené pouze pro detekci jedné konkrétní mutace. Tento přístup je tedy využíván pouze pro mutace s diagnostickým, prognostickým nebo terapeutickým významem a zároveň pokud se vyskytují u nádorů ve vysoké frekvenci. V případě mozkových nádorů je touto metodou detekována například mutace v genu BRAF nebo hot-spot mutace v IDH1 a IDH2 genech (11,12). Ve vyjmenovaných případech se jedná o mutace sloužící k přesné identifikaci nádoru, které mají vliv na prognózu a volbu léčby. Geny, které nemají „hot-spot“ mutace nebo geny, kde je koncentrováno více mutací v krátkém úseku, není možné touto metodou detekovat. Jedná se například o TP53 nebo CDKN2A (13,14). V těchto případech je k detekci mutací vhodné použít jiné molekulárně genetické metody, například sekvenování. Návrh sond pro detekci bodových mutací je obtížný zejména z důvodu nutnosti přesné lokalizace sondy, která musí končit v místě bodové mutace. Další komplikace je spojena se záměnou C→T nebo A→G. Nukleotidy G a T jsou schopny vytvářet vazby na principu slabých vodíkových můstků. Ligase-65 je stejně jako ostatní NAD dependentní ligázy méně citlivá k těmto záměnám a může dojít k falešně pozitivnímu signálu. V případě výskytu homologních genů, které by mohly zkreslit signál, je využit systém dvou ligací, tří sond následujících za sebou (obr. 3). Tento systém, pokud je navržen správně, dovoluje rozlišit mutace ve sledovaném genu od mutace v genu homologním.
Obr. 3. Využití dvou ligačního systému při detekce mutací (převzato z 2). 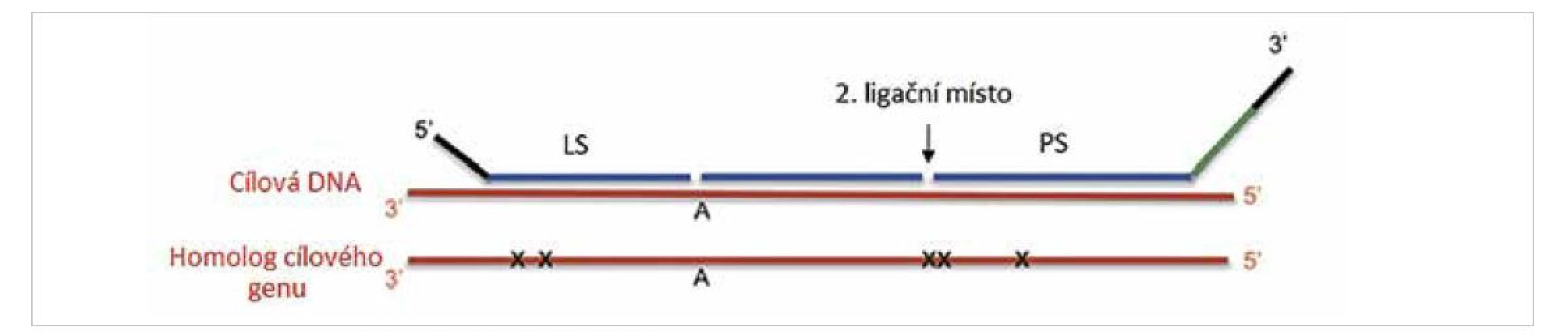
Stejně tak, jako u detekcí delecí/amplifikací je nutné při hodnocení přihlédnout k heterogenitě nádorové tkáně. Vzorky nádorů často obsahují směsi různých typů buněk, jako jsou stromální a/nebo imunitní buňky, nádorové buňky, zdravé buňky okolní tkáně. Při analýze bodových mutací je tedy nutné vzít tento fakt v úvahu. Bodové mutace lze spolehlivě detekovat, pokud je množství buněk s heterozygotní mutací 10 % až 15 % z celé buněčné populace (9). Tento detekční limit je srovnatelný s konvenčním (Sangerovym) i masivně paralelním sekvenováním.
HODNOCENÍ METYLACE POMOCÍ MLPA ANALÝZY
Při hodnocení nádorové DNA je kladen důraz nejen na genetické změny, ale i na změny epigenetické. Klíčovou roli v tomto procesu hraje hypermetylace promotorových oblastí tumor supresorových genů (15). Míru hypermetylace promotorů společně se změnami v počtu kopií genů je možné sledovat pomocí modifikace klasické MLPA reakce. Při této reakci je klíčovým krokem použití metylačně sensitivního restrikčního enzymu HhaI. Použití tohoto enzymu umožňuje rychlou, semikvantitativní detekci metylace. Metylačně specifická MLPA může být navržena pouze do hot-spot míst, která obsahují sekvenci GCGC specifických pro restrikční endonukleázu HhaI. Vlastním provedením se metylačně specifická MLPA neliší od standardní s jedinou výjimkou. Po skončení ligace jsou vzorky rozděleny do dvou reakcí. Jedna se provádí v přítomnosti HhaI v kombinaci s ligázou, druhý alikvot pouze s ligázou (obr. 4). Pokud není sekvence specifická pro HhaI restrikční endonukleázu u pacienta metylována, dojde k rozštěpení systému sonda/DNA vzorek a PCR templát je tím zničen. Pokud je ale cílová sekvence metylována, nedojde k jejímu rozpoznání restrikční endonukleázou HhaI, tím nedojde ke zničení PCR templátu a ten může být následně amplifikován při PCR kroku. Při hodnocení míry metylace tedy dochází k porovnání alikvotu ošetřeného restrikční endonukleázou a alikvotu, který enzym neobsahoval. Hlavní výhodou MLPA postupu je vynechání bisulfitové konverze nemetylovaných cytosinů, která je velmi obtížně standardizovatelná a zároveň je nutná u jiných molekulárně genetických metod, testujících metylační status (např. metylačně-specifická PCR nebo bisulfitové sekvenování). Tato metoda je vhodná i pro detekce metylace u mozkových nádorů (např. gliomů) a její citlivost je srovnatelná s metylačně specifickým PCR. Metylační status je správně identifikován u tkání, které obsahují 30 % metylované DNA. Výhodou MS MLPA testu je, kromě nepřítomnosti bisulfitové konverze, i možnost kombinace s CNV detekcí či detekcí bodových mutací v jedné reakci (2). Další výhodou je opět detekce až 50 sond v jedné reakci.
Obr. 4. Princip metylačně specifické MLPA reakce (převzato z 2). 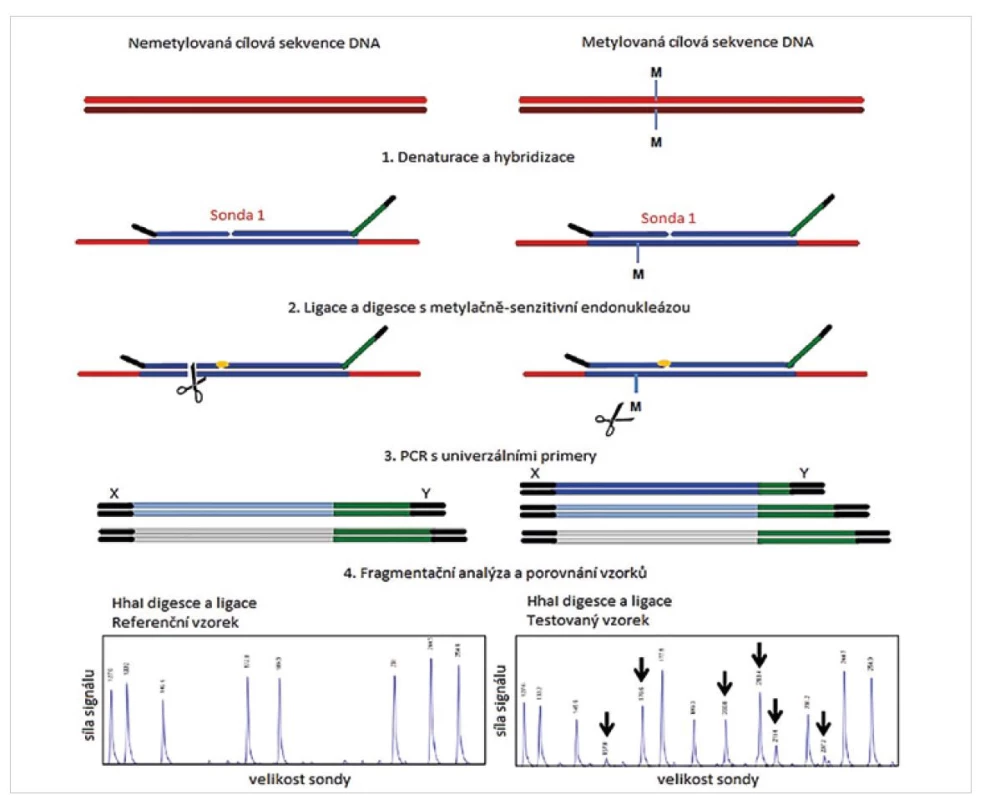
KORELACE MLPA A FISH/CISH V DIAGNOSTICE NÁDORŮ
Detekce mutací, delecí i amplifikací v nádorových tkáních je v současnosti prováděna pomocí více metod. Mezi rutinní a velmi spolehlivé patří například FISH (fluorescenční in-situ hybridizace) nebo jí příbuzná metodika CISH (chromogenní in-situ hybridizace) (16,17). Limitem obou hybridizačních metod je rozlišení, které je limitováno 20 kb. Všechny změny menší, než tento limit není možné detekovat. Hybridizační metodiky jsou také poměrně pracné, vyžadují dobře zapracované laboratorní pracovníky a jsou obtížně nastavitelné pro vyšší počet vzorků v laboratoři. Výhodou MLPA reakce je i možnost detekce více genetických markerů v jedné reakci, což hybridizační metody často neumožňují. Velikou výhodou MLPA analýzy je rychlost provedení při zachování citlivosti detekce. Obrázek 5 znázorňuje rozdílnou schopnost vybraných molekulárně genetických metod v detekci genetických změn. V neposlední řadě je nutné vyzdvihnout její všestrannost, lze ji použít k detekci změn počtu kopií jak u chromozomů, tak i u jednotlivých exonů sledovaných genů. Zároveň se jedná o metodu dostatečně citlivou k odlišení aberace v genech způsobujících onemocnění od velmi podobných pseudogenů.
Obr. 5. Využití MLPA reakce v molekulární diagnostice (převzato ze 7). 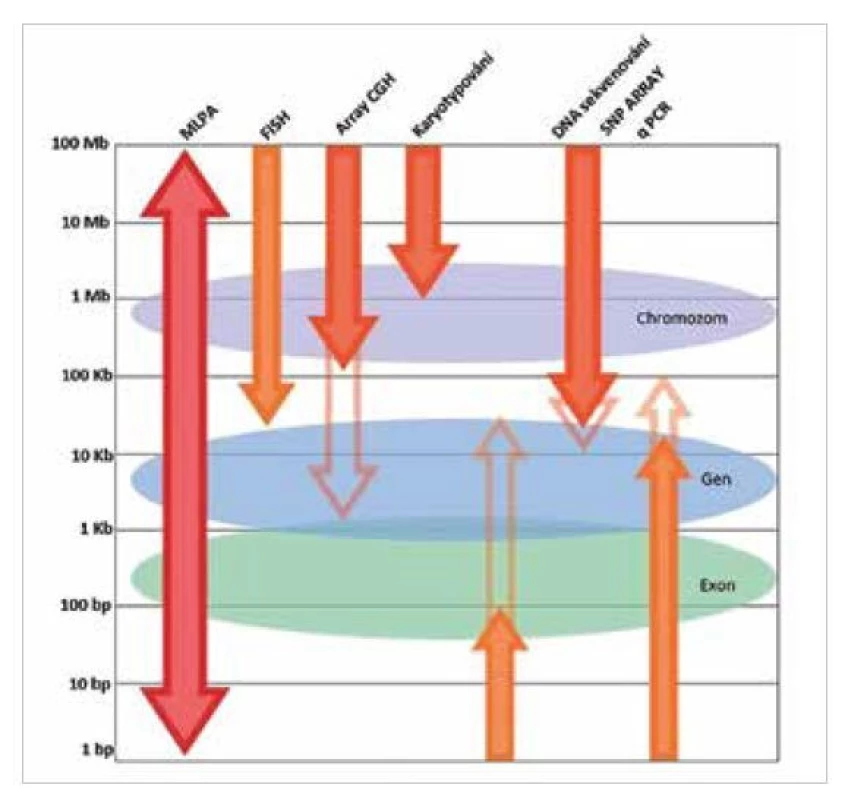
MOLEKULÁRNÍ DIAGNOSTIKA GLIOMŮ
Difuzní gliomy (DGs) jsou nádory centrálního nervového systému (CNS) charakterizované rozsáhlou infiltrací jednotlivými nádorovými buňkami s typickými cytologickými a histologickými obrazy gliové diferenciace (18). Tyto nádory postihují pacienty všech věkových kategorií a vznikají v celé CNS. Nejčastěji se však vyskytují u starších dospělých, nejběžnější lokalizací jsou mozkové hemisféry. Nejčastěji se v populaci vyskytují astrocytomy, méně často pak oligodendrogliomy a ependymomy a vzácně také gangliogliomy. Léčba zahrnuje resekci nádoru v co největším možném rozsahu, radioterapii i chemoterapii v závislosti na typu a gradu nádoru. Prognóza pacientů s gliomy je špatná, medián přežití se pohybuje okolo 15 měsíců (19). Jedním z nejagresivnějších nádorů u lidí je glioblastom. Původní klasifikace gliomů se odvíjela od morfologických znaků nádorových buněk, v posledních letech došlo k revidování WHO klasifikace s velkým důrazem na molekulární markery. Tento trend vedl k vydání 5. verze WHO klasifikace zahrnující molekulární markery jako plnohodnotné do klinické praxe (20).
Vývojem molekulárně genetického schématu pro testování gliových nádorů se zabývala mezinárodní multidisciplinární skupina, která vycházela z odpovědí na následující otázku: „Které molekulárně genetické markery jsou nutné pro klinicky relevantní diagnostiku difuzních gliomů u pacientů?“ (21).
Při výběru výsledných molekulárně genetických markerů se vycházelo z klinických studií splňujících následující kritéria: studie s lidskými pacienty; systematický přehled s/bez metaanalýzy dat, randomizovaná klinická studie, prospektivní nebo retrospektivní design studie, minimálně 30 pacientů ve studii (s odůvodněnými výjimkami – například nádory dětského věku), studie publikována v angličtině; studie zahrnující měřitelné údaje.
Sledovány byly následující genetické a molekulární markery: změny genů IDH1/2, H3, BRAF, ATRX, TP53, MYB, MYBL1, TERT, FGFR, EGFR, PDGFRA, MET, CDKN2A, PTEN, NF1, MGM2 a CDK4 a dále: 1 p/19 q kodelece, zmnožení chromozomu 7, ztráty chromosomu 10, metylace/mutace promotoru MGMT genu, mikrosatelitová instabilita. Výsledkem rozsáhlé studie jsou schémata na obrázcích 6 a 7, které zobrazují diferenciální diagnostiku zohledněnou o molekulárně genetickou diagnostiku.
Obr. 6. Schéma diagnostiky IDH mutovaných gliomů (převzato z 21). 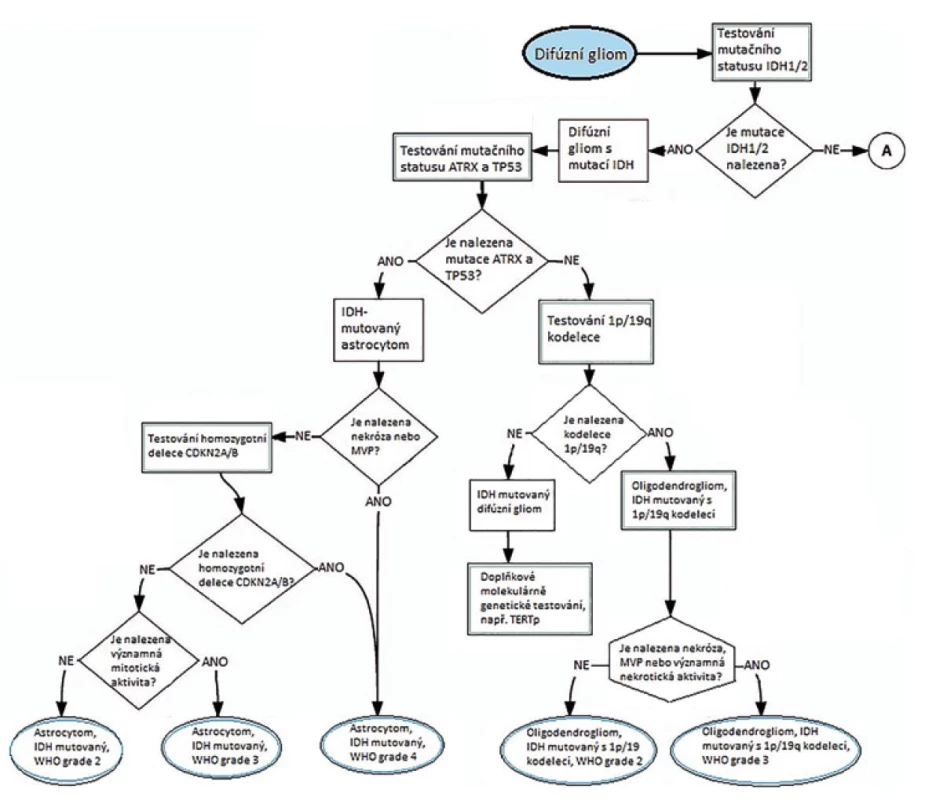
Obr. 7. Diagnostika gliových nádorů vázaná na výsledky molekulárně patologické vyšetření (převzato z 21). 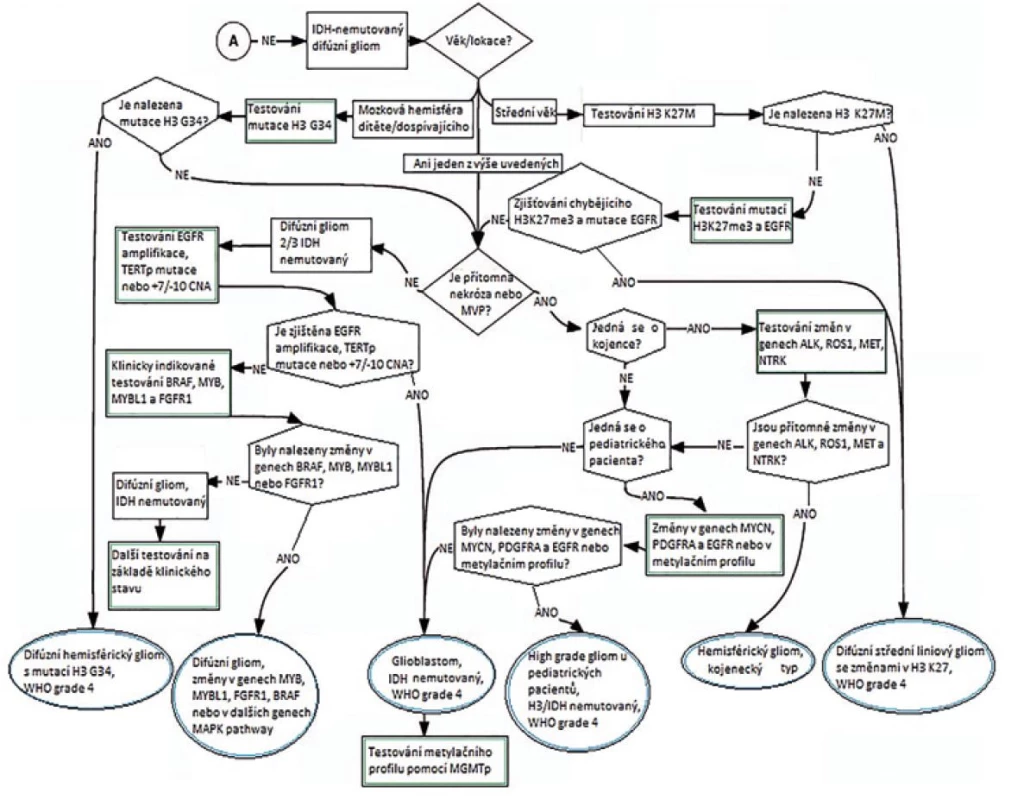
PŘÍKLADY MLPA ANALÝZY ZÍSKANÉ V MOLEKULÁRNĚ PATOLOGICKÉ LABORATOŘI KNL, A. S. A V LABORATOŘI BIOLOGIE SOLIDNÍCH TUMORŮ 2. LF UK A FNM
Následující kapitola popisuje typické MLPA nálezy u pacientů. Laboratoř molekulární patologie rutinně využívá všechny dostupné MLPA systémy vybrané pro diagnostiku mozkových nádorů. Konkrétně se jedná o SALSA MLPA Probemix P105 Glioma-2, SALSA MLPA Probemix P088 Oligodendroglioma 1p-19q, SALSA MLPA Probemix P370 BRAF-IDH1-IDH2 a SALSA MLPA Probemix ME012 MGMT-IDH1-IDH2 (7).
Probemix P105 obsahuje 56 testovacích sond a slouží k detekci genetických změn u genů PDGFRA (4q12), EGFR (7p11.2), CDKN2A (9p21.3), PTEN (10q23.31), CDK4, MIR26A2, MDM2 (12q14-q15), NFKBIA (14q13.2) a TP53 (17p13.1). Nález somatické mutace nebo delece/amplifikace ve třech následujících kritických signálních drahách predikuje rozvoj gliomu v testované tkáni, a) dráha RTK/ PI3K (zahrnující např. geny EGFR, PDGFRA a PTEN), b) dráha p53 (zahrnující např. geny CDKN2A, MDM2 a TP53) c) dráha RB (zahrnující např. geny CDKN2A a CDK4). Nejčastěji nacházené výsledky jsou prezentovány na obrázcích 8, 9 a 10.
Obr. 8. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix P105 Glioma-2. Typický obraz amplifikace genu EGFR bez možnosti hodnocení dalších změn. Výrazná amplifikace genu EGFR znemožňuje hodnocení signálu dalších sond. 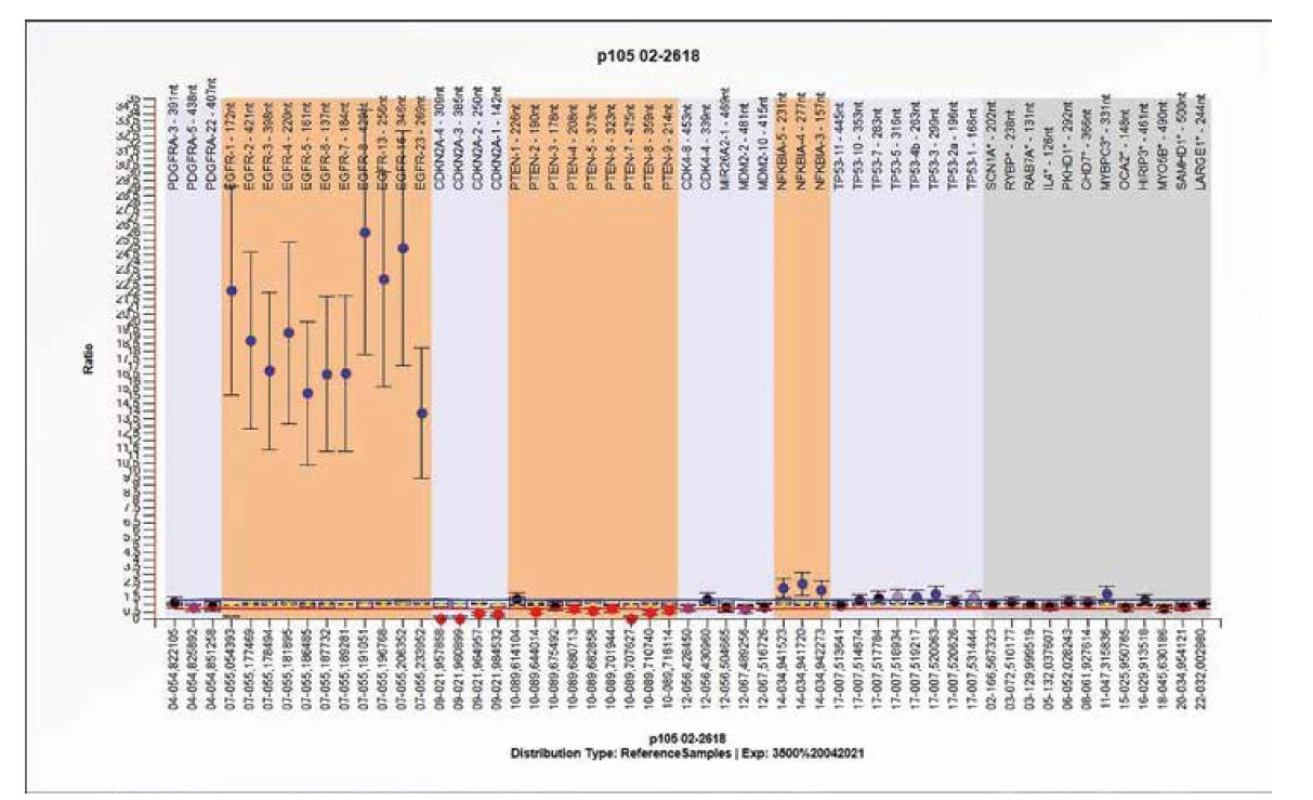
Obr. 9. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix P105 Glioma-2. Typický obraz amplifikace genu EGFR s amplifikací genu NFKBIA. 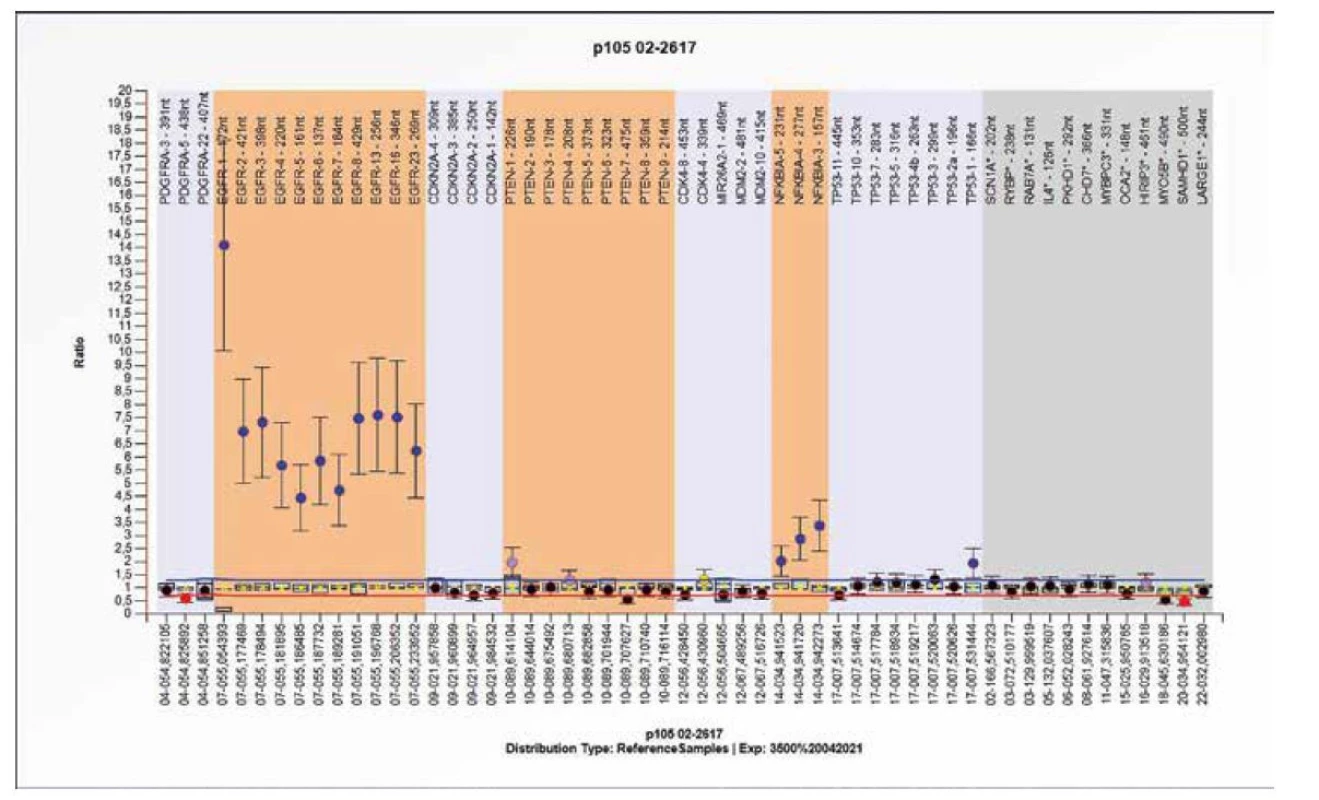
Obr. 10. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix P105 Glioma-2. Typický obraz zmnožení (gain) EGFR genu s delecí CDKN2A genu. 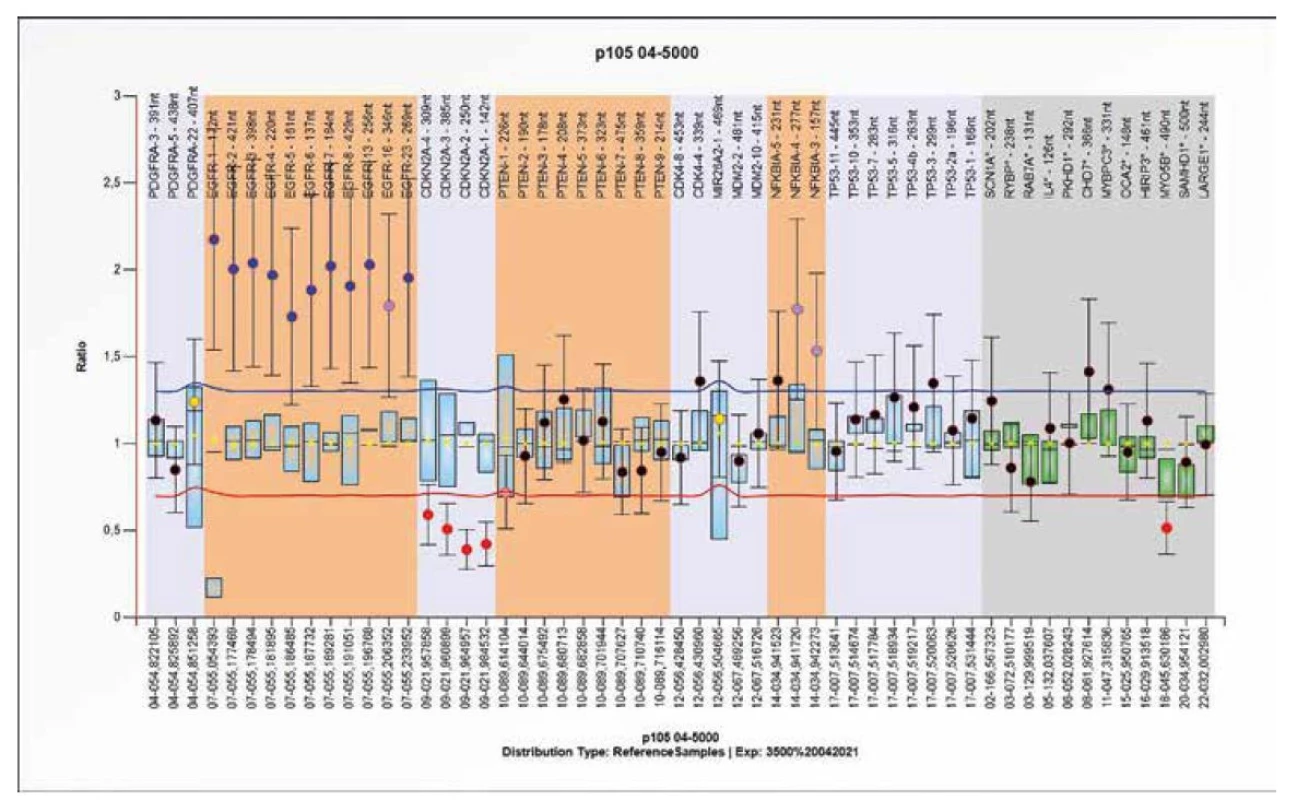
Probemix P088 obsahuje 59 sond, slouží k detekci změn počtu kopií na krátkých raménkách prvního chromozomu a dlouhých raménkách 19 chromozomu. Dále lze detekovat delece CDKN2A a CDKN2B genů a čtyři nejběžnější somatické bodové mutace v genech IDH1 (p.R132C a p.R132H) a IDH2 (p.R172K a p.R172M). Ostatní bodové mutace přítomné v IDH1/2 genech nejsou detekovány. Nález bodové mutace pomocí MLPA sondy by měl být ověřen jinou molekulárně genetickou metodou. Následující obrázky 11 a 12 zobrazují typické nálezy získané pomocí probemixu P088.
Obr. 11. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix P088 Oligodendroglioma. Výsledek získaný analýzou DNA z FFPE bločku. Detekována je delece CDKN2A a CDKN2B genů a dále R172M mutace IDH2 genu. 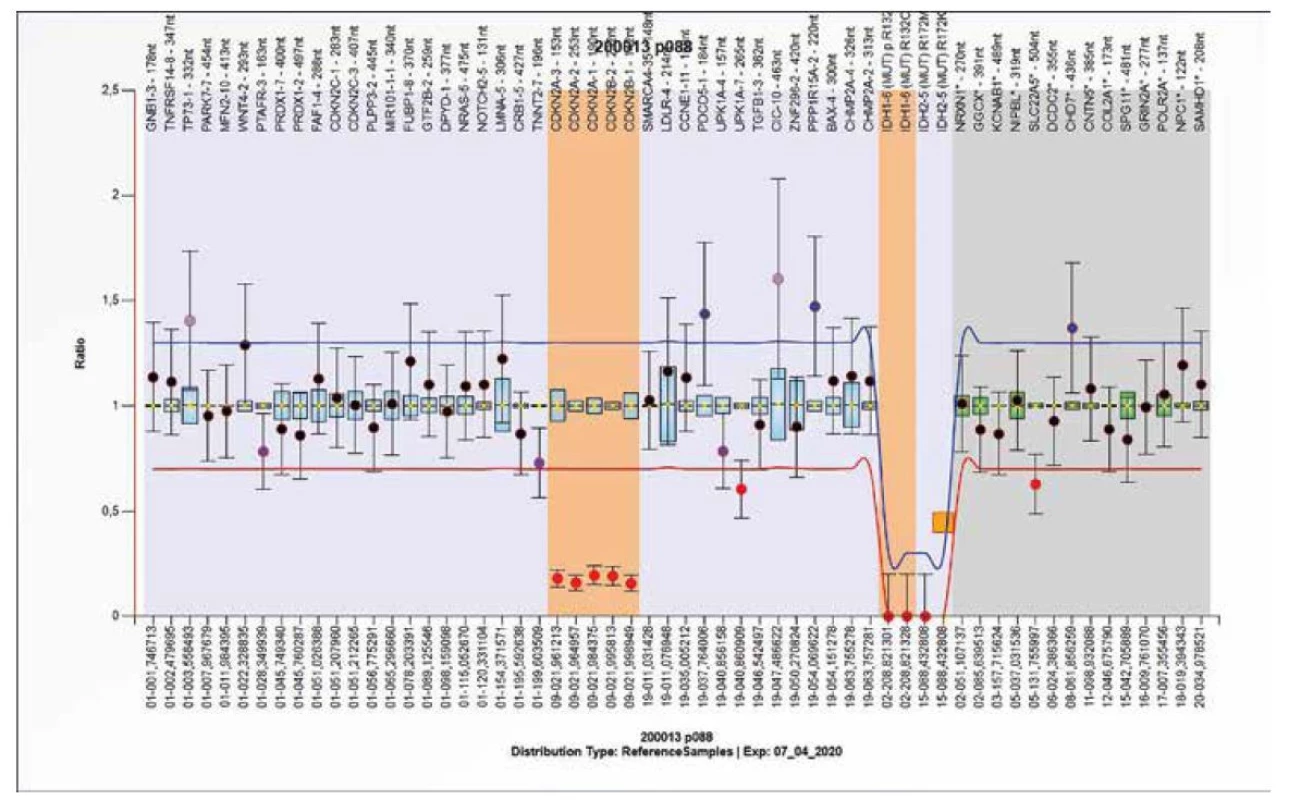
Obr. 12. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix P088 Oligodendroglioma. Typický obraz kodelece 1p/19q a mutace R132C v IDH1 genu. 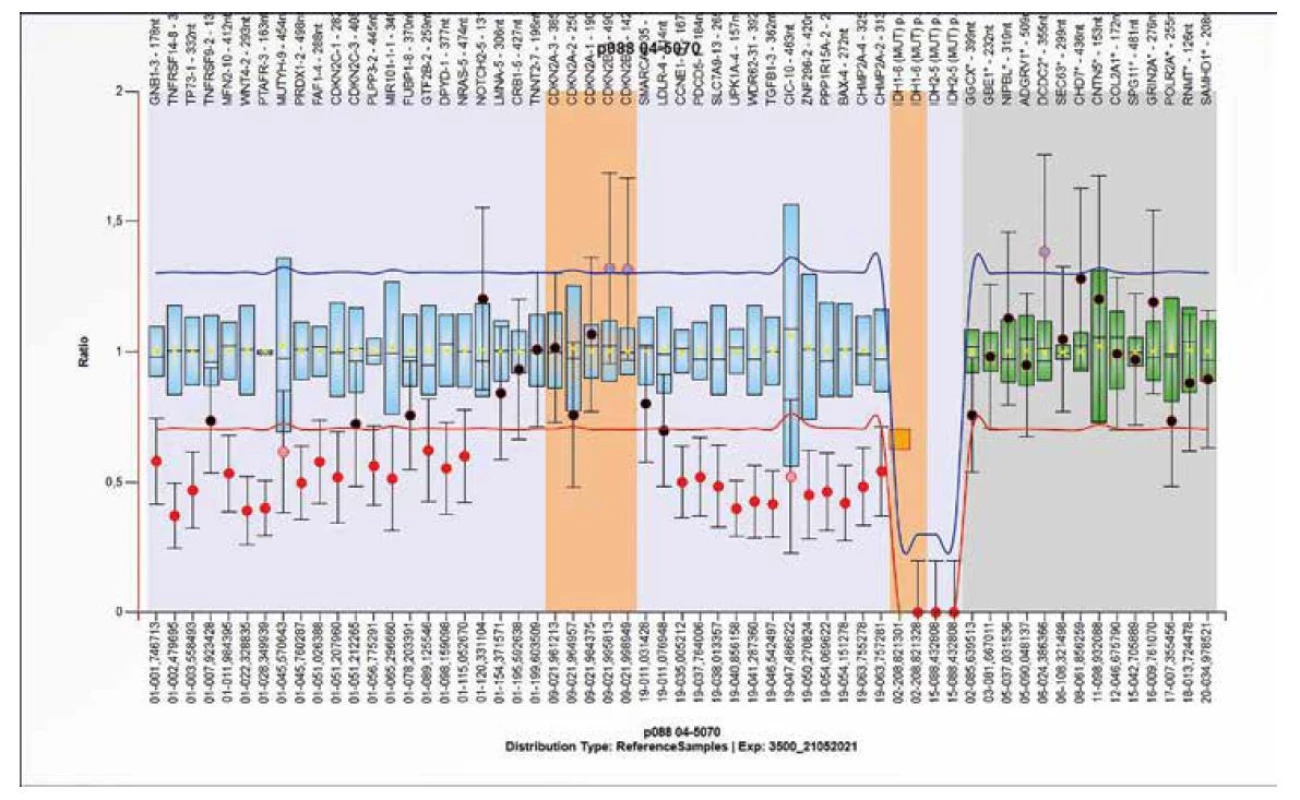
SALSA MLPA Probemix P370 BRAF-IDH1-IDH2 je test sloužící k detekci BRAF p.V600E a čtyř nejčastějších mutací IDH1 (p.R132H a p.R132C) a IDH2 (p.R172M a p.R172K) genů (viz obrázek 13). Dále detekuje genomové duplikace vedoucí k fúzním genům SRGAP3-RAF1, KIAA1549-BRAF a FGFR1-TACC1 na chromozomu 3p, 7q a 8p. V neposlední řadě slouží k detekci duplikace/ delece v BRAF, CDKN2A /2B, FGFR1, MYB a MYBL1 genech. Aktivace MAPK dráhy (vznik fúzního genu KIAA1549-BRAF na 7q34) je typická pro pilocytární astrocytomy (22). Ukázka BRAF mutovaného vzorku je na obrázku 13.
Obr. 13. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix P370 BRAF-IDH1-IDH2. Výsledek záchytu bodové mutace V600E v genu BRAF. 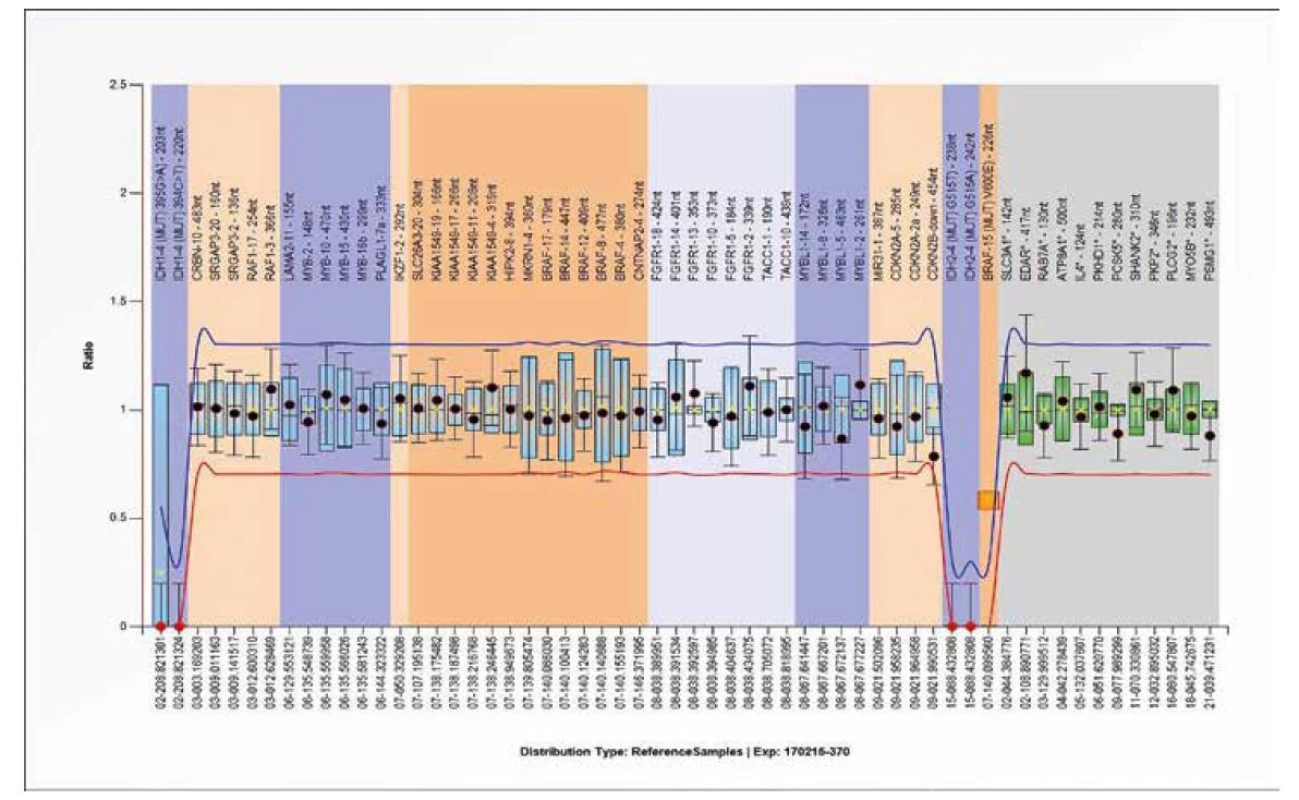
SALSA MS-MLPA Probemix ME012 MGMT-IDH1-IDH2 je test sloužící k detekci aberantní metylace genu MGMT. Tento probemix lze také použít k detekci přítomnosti bodových mutací v genech IDH1 (p.R132H=c.395G>A a p.R132C=c.394C>T) a IDH2 (p.R172K=c.515G>A a p.R172M =c.515G>T).
Hypermetylace v promotorové oblasti genu MGMT, kódující enzym opravující DNA O6-methylguanin DNA methyltransferázu, je důležitým prognostickým markerem a prediktorem odpovědi na léčbu u gliomu alkylačními činidly, jako je například temozolomid (23,24,25). Nález mutací v IDH1/2 genech společně s metylací MGMT promotoru slouží jako prediktor přežití pacientů s glioblastomem (26). Detekce mutací IDH1/2 genu společně s hypermetylací MGMT by mělo znamenat lepší prognózu než samotná IDH1/2 mutace nebo MGMT hypermetylace (27). Ukázky delece a zvýšené metylace genu MGMT jsou znázorněné na obrázcích 14 a 15.
Obr. 14. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix ME012 MGMT-IDH1-IDH2. Typický obraz delece MGMT genu ve sledovaných oblastech. 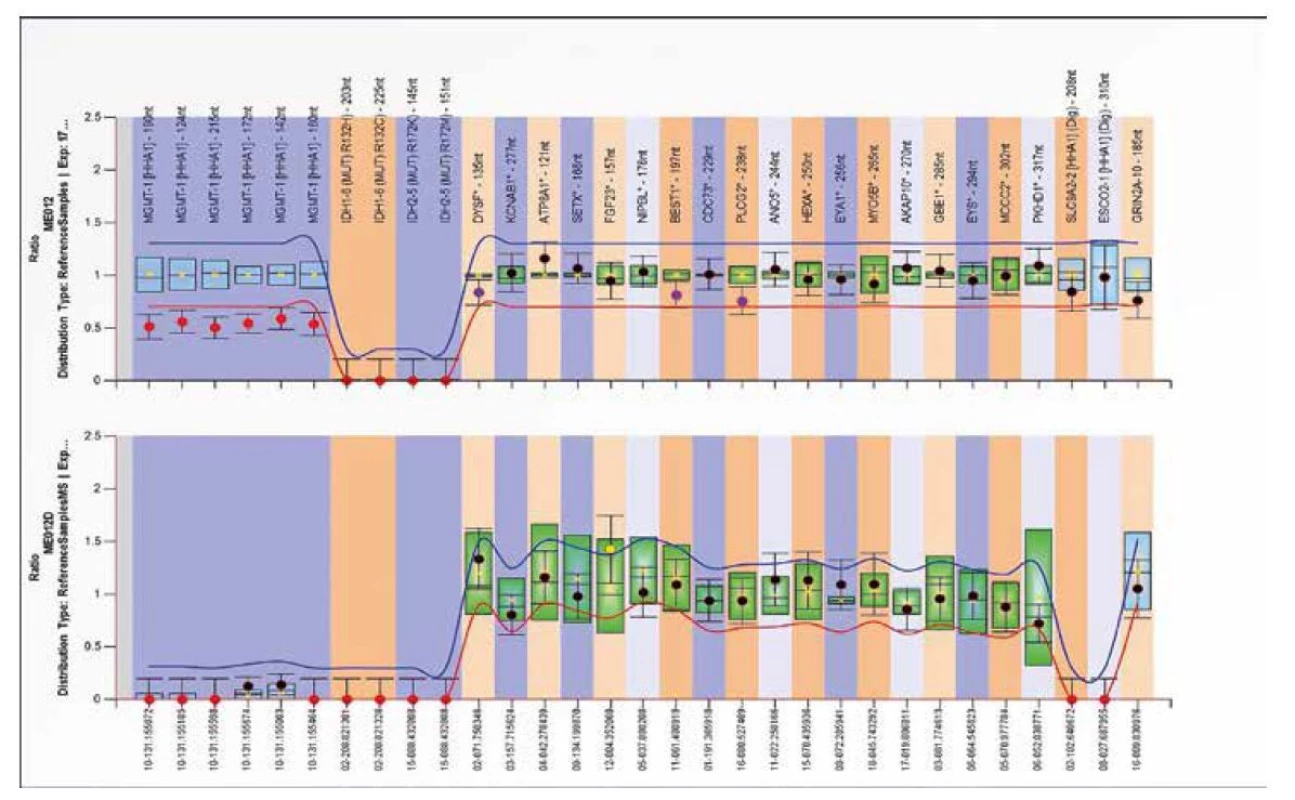
Obr. 15. Výsledek molekulárně genetického vyšetření tkáně pomocí SALSA MLPA Probemix ME012 MGMT-IDH1-IDH2. Typický obraz zvýšené metylace MGMT genu ve sledovaných oblastech 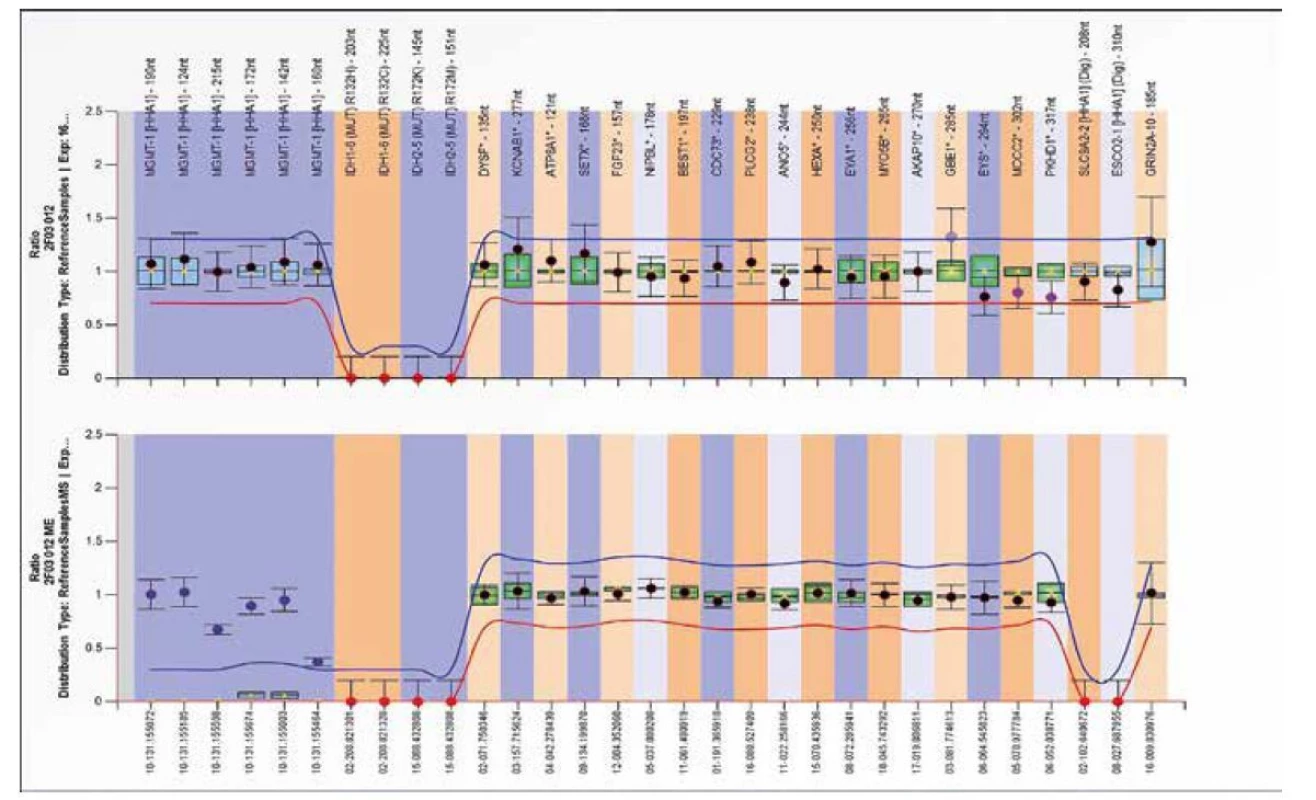
PROHLÁŠENÍ
Autor práce prohlašuje, že v souvislosti s tématem, vznikem a publikací tohoto článku není ve střetu zájmů a vznik ani publikace článku nebyly podpořeny žádnou farmaceutickou firmou. Toto prohlášení se týká i všech spoluautorů.
Adresa pro korespondenci:
Mgr. Iva Dolinová, Ph.D.
Oddělení molekulární genetiky a diagnostiky, Centrum PATOS
Husova 357/10, 460 63, Liberec
tel: 485 313 008
e-mail: iva.dolinova@nemlib.cz
Zdroje
1. Schouten JP, Cathal JM, Waaijer R, Zwijnenburg D, Diepvens F, Pals G. Relative quantification of 40 nucleic acid sequences by multiplex ligation-dependent probe amplification. Nucleic Acids Res 2002; 30(12): 57.
2. Hömig-Hölzel C, Savola S. Multiplex ligation - dependent probe amplification (MLPA) in tumor diagnostics and prognostics. Diagn Mol Pathol 2012; 21(4): 189-206.
3. Kozlowski P, Roberts P, Dabora S, et al. Identification of 54 large deletions/duplications in TSC1 and TSC2 using MLPA, and genotype - phenotype correlations. Hum Genet 2007; 121 : 389-400.
4. MRC-Holland. MS-MLPA General Protocol (One-Tube). 2020; dostupné na www.mlpa.cz.
5. Troxler H, Kleinert P, Schmugge M, Speer O. Advances in hemoglobinopathy detection and identification. Advances in clinical chemistry 2012; 57 : 2.
6. Sellner LN, Taylor GR. MLPA and MAPH: New Techniques for Detection of Gene Deletions. Hum Mutat 2004; 23 : 413-419.
7. MRC-Holland. MLPA® General information. 2022; dostupné na www.mlpa.cz.
8. Tate JG, Bamford S, Jubb HC, et al. COSMIC: the catalogue of somatic mutations in cancer. Nucleic acids Res 2019; 47(D1): D941-D947.
9. Coffa J, Berg J. Analysis of MLPA data using novel softwere Coffalyser. NET by MRC-Holland. In: Eldin, A. B, ed. Modern Approaches to Quality Control. Rijeka, Croatia: In Tech. 2011;125-150.
10. Van Opstal D, Boter M, De Jong D, Van Den Berg C, Brüggenwirth HT, Wildschut HI, Galjaard RJH. Rapid aneuploidy detection with multiplex ligation-dependent probe amplification: a prospective study of 4000 amniotic fluid samples. Eur J Hum Genet 2001; 17(1): 112-121.
11. Sullivan RJ, Flaherty KT. BRAF in melanoma: pathogenesis, diagnosis, inhibition, and resistance. J Skin Cancer 2011. doi. org/10.1155/2011/423239.
12. Eckel-Passow JE, Lachance DH, Molinaro AM, et al. Glioma groups based on 1p/19q, IDH, and TERT promoter mutations in tumors. N Engl J Med 2015; 372(26): 2499-2508.
13. Reis GF, Pekmezci M, Hansen HM, et al. CDKN2A loss is associated with shortened overall survival in lower-grade (World Health Organization Grades II–III) astrocytomas. J Neuropathol E Neurol 2015; 74(5): 442-452.
14. Gillet E, Alentorn A, Doukouré B, et al. TP53 and p53 statuses and their clinical impact in diffuse low grade gliomas. J Neur Onco 2014; 118(1): 131-139.
15. Christensen BC, Smith AA, Zheng S, et al. DNA metylation, isocitrate dehydrogenase mutation, and survival in glioma. J Natl Cancer Inst 2011; 103(2):143-153.
16. Jha P, Sarkar C, Pathak P, Sharma MC, et al. Detection of allelic status of 1p and 19q by microsatellite-based PCR versus FISH: limitations and advantages in application to patient management. Diag Mol Pathol 2011; 20(1): 40-47.
17. Marquez A, Wu R, Zhao J, et al. Evaluation of Epidermal Growth Factor Receptor (EGFR) by Chromogenic In Situ: Hybridization (CISH™) and Immunohistochemistry (IHC) in Archival Gliomas Using Bright-Field Microscopy. Diag Mol Pathol 2004; 13(1): 1-8.
18. Louis DN, Perry A, Reifenberger G, et al. The 2016 World Health Organization classification of tumors of the central nervous system: a summary. Acta neuropathologica 2016; 131(6): 803-820.
19. Stupp R, Mason WP, van den Bent MJ, et al. Radiotherapy plus concomitant and adjuvant temozolomide for glioblastoma. N Engl J Med 2005; 352 : 987-996.
20. Louis DN, Peery A, Wesseling P, et al. The WHO classification of tumors of the central nervous system: a summary. Neur Oncol 2021; 23(8): 1231-1251.
21. Brat DJ, Aldape K, Bridge JA, et al. Molecular Biomarker Testing for the Diagnosis of Diffuse Gliomas. Arch Pathol Lab Med 2022. doi: 10.5858/arpa.2021-0295-CP.
22. Jones DT, Kocialkowski S, Liu L, et al. Tandem duplication producing a novel oncogenic BRAF fusion gene defines the majority of pilocytic astrocytomas. Cancer Res 2008; 68(21): 8673-8677.
23. Weller M., Felsberg J, Hartmann C, et al. Molecular predictors of progression-free and overall survival in patients with newly diagnosed glioblastoma: a prospective translational study of the German Glioma Network. J Clin Oncol 2009; 27(34): 5743-5750.
24. Hegi ME, Diserens AC, Gorlia T, et al. MGMT gene silencing and benefit from temozolomide in glioblastoma. N Engl J Med 2005; 352(10): 997-1003.
25. Pegg A E. Mammalian O6-alkylguanine-DNA alkyltransferase: regulation and importance in response to alkylating carcinogenic and therapeutic agents. Cancer Res 1990; 50(19): 6119-6129.
26. Wick W, Meisner C, Hentsch B, et al. Prognostic or predictive value of MGMT promoter methylation in gliomas depends on IDH1 mutation. Neurology 2013; 81(17): 1515-1522.
27. Molenaar RJ, Verbaan D, Lamba S, et al. The combination of IDH1 mutations and MGMT methylation status predicts survival in glioblastoma better than either IDH1 or MGMT alone. Neur Oncol 2014; 16(9): 1263 - 1273.
Štítky
Patológia Súdne lekárstvo Toxikológia
Článek 'NEUROPATOLOGIEČlánek 'NEFROPATOLOGIEČlánek 'HEPATOPATOLOGIEČlánek 'ORTOPEDICKÁ PATOLOGIEČlánek 'KARDIOPATOLOGIEČlánek 'HEMATOPATOLOGIEČlánek 'CYTODIAGNOSTIKAČlánek 'PATOLOGIE GITČlánek 'PATOLOGIE ORL OBLASTIČlánek 'PULMOPATOLOGIEČlánek 'UROPATOLOGIEČlánek Mukormykóza: kazuistika
Článok vyšiel v časopiseČesko-slovenská patologie

2022 Číslo 3-
Všetky články tohto čísla
- Novinky ve WHO klasifikaci nádorů CNS 2021
- … obor paleontologie se v roce 1989 otevíral jenom v Leningradě …
- 'NEUROPATOLOGIE
- 'NEFROPATOLOGIE
- 'HEPATOPATOLOGIE
- 'ORTOPEDICKÁ PATOLOGIE
- 'KARDIOPATOLOGIE
- 'HEMATOPATOLOGIE
- 'CYTODIAGNOSTIKA
- 'PATOLOGIE GIT
- 'PATOLOGIE ORL OBLASTI
- 'PULMOPATOLOGIE
- 'UROPATOLOGIE
- Novinky ve WHO klasifikaci nádorů centrálního nervového systému 2021
- Efektivní schéma využívané v diagnostice nádorů CNS
- Molekulárně patologické profilování vybraných nádorů centrálního nervového systému metodou MLPA
- Nádory CNS – klinické a radiologické aspekty
- Obrovskobuněčný fibroblastom: kazuistika
- Mukormykóza: kazuistika
- Česko-slovenská patologie
- Archív čísel
- Aktuálne číslo
- Informácie o časopise
Najčítanejšie v tomto čísle- Novinky ve WHO klasifikaci nádorů centrálního nervového systému 2021
- Nádory CNS – klinické a radiologické aspekty
- Mukormykóza: kazuistika
- Obrovskobuněčný fibroblastom: kazuistika
Prihlásenie#ADS_BOTTOM_SCRIPTS#Zabudnuté hesloZadajte e-mailovú adresu, s ktorou ste vytvárali účet. Budú Vám na ňu zasielané informácie k nastaveniu nového hesla.
- Časopisy



